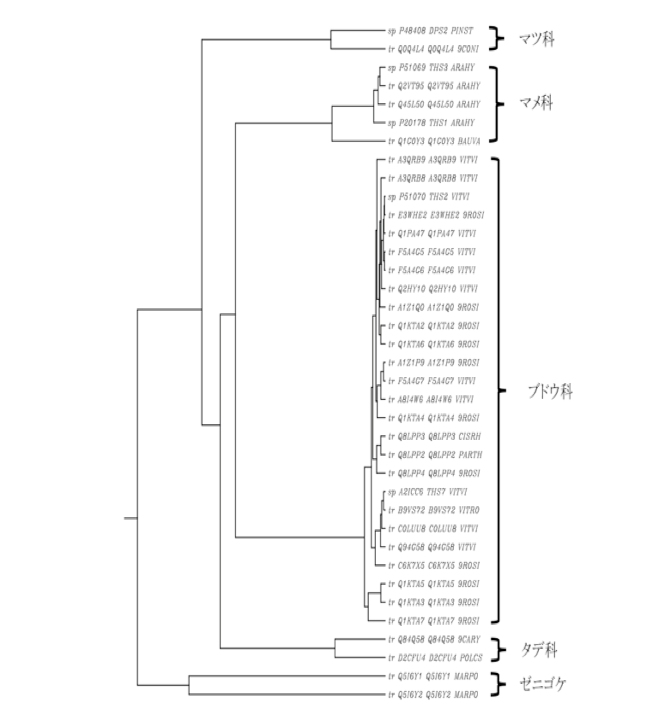
たどたどしい解析に用いた被子植物カルコンシンターゼのアミノ酸配列を列記しておく。奇特な人がこれらを用いてもっと精密な解析をし、より良い結果を出してくれれば最高である。もちろん、この解析をしてから8年ほどの時間が経っていることを考えれば、使える配列の数は倍増しているに違いない。今からお前がやればいいじゃないかと言われるかもしれないが、この結果は私にとって一つの通過点に過ぎない。書きたいことはまだたくさん残っている。もうすぐ後期高齢者に手が届くようになったー後期高齢者:嫌な言葉だね、愛情がないよ、筆頭老中で行きましょうー私にとって、再解析に使う時間があったら別のことを語りたい。ふふふ、騙りたいが本音かもしれない。
解析に使用した被子植物カルコンシンターゼのアミノ酸配列
>sp|P30074|CHS2_MEDSA Chalcone synthase 2 OS=Medicago sativa GN=CHS2 PE=1 SV=1
MVSVSEIRKAQRAEGPATILAIGTANPANCVEQSTYPDFYFKITNSEHKTELKEKFQRMCDKSMIKRRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPRLGKEAAVKAIKEWGQPKSKITHLIVCTTSGVDMPGADYQLTKLLGLRPYVKRYMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEVTAVTFRGPSDTHLDSLVGQALFGDGAAALIVGSDPVPEIEKPIFEMVWTAQTIAPDSEGAIDGHLREAGLTFHLLKDVPGIVSKNITKALVEAFEPLGISDYNSIFWIAHPGGPAILDQVEQKLALKPEKMNATREVLSEYGNMSSACVLFILDEMRKKSTQNGLKTTGEGLEWGVLFGFGPGLTIETVVLRSVAI
>sp|P13114|CHSY_ARATH Chalcone synthase OS=Arabidopsis thaliana GN=CHS PE=1 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>sp|P23569|CHSY_PUEML Chalcone synthase OS=Pueraria montana var. lobata GN=CHS PE=1 SV=1
MVSVAEIRQAQRAEGPATILAIGTANPPNCVDQSTYPDYYFRITNSEHMTELKEKFQRMCDKSMIKKRYMYLTEEILKENPNMCAYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLIFCTTSGVDMPGADYQLTKQLGLRPYVKRYMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIVGSDPIPQVEKPLYELVWTAQTIAPDSEGAIDGHLREVGLTFHLLKDVPGIVSKNIDKALFEAFNPLNISDYNSIFWIAHPGGPAILDQVEQKLGLKPEKMKATRDVLSDYGNMSSACVLFILDEMRRKSAENGLKTTGEGLEWGVLFGFGPGLTIETVVLRSVAI
>sp|Q9FUB7|CHSY_HYPAN Chalcone synthase OS=Hypericum androsaemum PE=1 SV=1
MVTVEEVRKAQRAEGPATVMAIGTAVPPNCVDQATYPDYYFRITNSEHKAELKEKFQRMCDKSQIKKRYMYLNEEVLKENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAAIIIGSDPIPEVEKPLFELVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNVEKSLTEAFKPLGISDWNSLFWIAHPGGPAILDQVEAKLSLKPEKLRATRHVLSEYGNMSSACVLFILDEMRRKSKEDGLKTTGEGIEWGVLFGFGPGLTVETVVLHSVAIN
>sp|Q8RVK9|CHS_CANSA Naringenin-chalcone synthase OS=Cannabis sativa GN=CHS PE=1 SV=1
MVTVEEFRKAQRAEGPATIMAIGTATPANCVLQSEYPDYYFRITNSEHKTELKEKFKRMCDKSMIRKRYMHLTEEILKENPNLCAYEAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGSAALIVGSDPIPEVEKPIFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLNEAFKPLGISDWNSLFWIAHPGGPAILDQVESKLALKTEKLRATRHVLSEYGNMSSACVLFILDEMRRKCVEDGLNTTGEGLEWGVLFGFGPGLTVETVVLHSVAI
>tr|B5LXY1|B5LXY1_GOSHI Chalcone synthase OS=Gossypium hirsutum GN=CHS5 PE=2 SV=1
MVTVEEVRKAQRAQGPATVLAIGTSTPPNCVDQSTYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIIGADPMPEIEKPMFEIVSVAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGISDWNSLFWIAHPGGPAILDQVEAKLALKPEKLRATRHVLSEYGNMSSACVLFILDEMRKKSREDGLQTTGEGLEWGVLFGFGPGLTVETVVLHSVAA
>tr|A7L2Z4|A7L2Z4_GOSHI Chalcone synthase OS=Gossypium hirsutum GN=CHS PE=2 SV=1
MVTVEEVRKAQRAQGPATVLAIGTSTPPNCVDQSTYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIIGADPVPEIEKPMFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGISDWNSLFWIAHPGGPAILDQVEAKLALKPEKLRATRHVLSEYGNMSSACVLFILDEMRKKSREDGLQTTGEGLEWGVLFGFGPGLTVETVVLHSVAA
>tr|G3G7Y9|G3G7Y9_GOSHI Chalcone synthase OS=Gossypium hirsutum GN=CHS3 PE=2 SV=1
MATVEEIRKALRAQGPATVLAIGTATLPNCVFQADYPDYYFRITNSDHMTDLKHKFKRMCDKSMIKKRHMYLTEEILKENPNMCAYMASSLDARQDIVVVEVPKLGKEAATKAIKEWGHPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRIMMYQQGCFAGGTVLRLAKDLAENNKDARVLVVCSEITAVTFRGPSDTHLDSLVGQALFADGAGAVIIGADPDSKTERPLYQFVSAAHTILPDSDGAIDGHLREVGLNFHLLKDVPGLISKNIEKSLVEAFSPIGIWDWNSIFWIAHPGGPAILDQIEAKLGLKEDKLRATRHVLSEFGNMSSACVLFIMDEMRKKSLDQGMPTTGEGYEWGVLFGFGPGLTVETVVLHSIPTRAN
>tr|G3G7Z2|G3G7Z2_GOSHI Chalcone synthase OS=Gossypium hirsutum GN=CHS6 PE=2 SV=1
MVTVEEVRKAQRAQGPATVLAIGTSTPPNCVDQSTYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMFLTEEILKENPNVCAYMEPSLDARQDMVVVEVPKLGKEAAARAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNEGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIIGSDPIPEIEKPLFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGITDWNSLFWIAHPGGPAILDQVEAKLALKPEKLRATRHVLSEYGNMSSACVLFILDEMRNKSREDGVQTTGEGLEWGVLFGFGPGLTVETVVLHSIPA
>tr|C8YQU8|C8YQU8_GOSHI Chalcone synthase 1 OS=Gossypium hirsutum PE=2 SV=1
MVTVEEVRKAQRAQGPATVLAIGTSTPPNCVDQSTYPDYYFHITNSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIIGADPMPEIEKPMFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGISDWNSLFWIAHPGGPAILDQVEAKLALKPEKLRATRHVLSEYGNMSSACVLFILDEMRKKSREDGLQTTGEGLEWGVLFGFGPGLTVETVVLHSVAA
>tr|G3G7Y8|G3G7Y8_GOSHI Chalcone synthase OS=Gossypium hirsutum GN=CHS2 PE=2 SV=1
MVTVEEVRKAQRAEGPATVLAIGTSTPPNCVDQSTYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPRLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIVGADPLSEIEKPMFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLAEAFQPLGISDWNSLFWIAHPGGPAILDQVEAKLALKPEKLRATRHVLSEYGNMSSACVLFILDEMRKKSKEDGLGTTGEGLEWGVLFGFGPGLTVETVVLHSISA
>tr|B7SQE0|B7SQE0_ABEMA Chalcone synthase OS=Abelmoschus manihot GN=CHS PE=2 SV=1
MVTVEEVRKAQRAEGPATVLAIGTSTPPNCVDQSTYPDYYFRITKSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIIGADPIPEIEKPMFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGISDWNSLFWIAHPGGPAILDQVEAKLALKPEKLRATRHVLSEYGNMSSACVLFILDEMRKSSKENGLGTTGEGLEWGVLFGFGPGLTVETVVLHSVTA
>tr|A0AMG9|A0AMG9_HUMLU Naringenin-chalcone synthase OS=Humulus lupulus GN=chs_H1-211 PE=2 SV=1
MVTVEEVRKAQRAEGPATILAIGTATPANCILQSEYPDYYFRITNSEHKTELKEKFKRMCDKSMIRKRYMHLTEEILKENPNLCAYEAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGSAALIIGADPIPEIEKPIFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFKPLGISDWNSLFWIAHPGGPAILDQVESKLALKPEKLRATRHVLGEYGNMSSACVLFILDEMRRKCAEDGLKTTGEGLEWGVLFGFGPGLTVETVVLHSVGI
>tr|B9VI85|B9VI85_HUMLU Naringenin-chalcone synthase OS=Humulus lupulus PE=2 SV=1
MVTVEEVRKAQRAEGPATILAIGTATPANCILQSEYPDYYFRITNSEHKTELKEKFKRMCDKSMIRKRYMHLTEEILKENPNLCAYEAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLGSLVGQALFGDGSAALIIGADPIPEIEKPVFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFKPLGISDWNSLFWIAHPGGPAILDQVESKLALKPEKLRATRHVLGEYGNMSSACVLFILDEMRRKCAEDGLKTTGEGLEWGVLFGFGPGLTVETVVLHSVGI
>tr|A0AMG8|A0AMG8_HUMLU Naringenin-chalcone synthase OS=Humulus lupulus GN=chs_H1-132 PE=2 SV=1
MVTVEEVRKAQRAEGPATILAIGTATPANCILQSEYPDYYFRITNSEHKTELKEKFKRMCDKSMIRKRYMHLTEEILKENPNLCAYEAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRVAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGSAALIIGADPTPEIEKPIFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFKPLGISDWNSLFWIAHPGGPAILDQVESKLALKPEKLRATRHVLGEYGNMSSACVLFILDEMRRKCAEDGLKTTGEGLEWGVLFGFGPGLTVETVVLHSVGI
>sp|O82144|CHSY_HYDMC Chalcone synthase OS=Hydrangea macrophylla GN=CHS PE=2 SV=1
MVTVEEVRKAQRAEGPATILAIGTATPPNYVDQSTYPDFYFRVTNSEHKKELKAKFQRMCDNSQIKKRYMHLTEEILKENPNICAYMAPSLDARQDMVVVEIPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIIGSDPMPEVEKPLFEIVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFRPLDISDWNSIFWIAHPGGPAILDQVEKKLALKPEKLRATRNVLSDYGNMSSACVLFIMDEMRKNSAEEGLMTTGEGLEWGVLFGFGPGLTVETVVLHGVST
>tr|O82145|O82145_HYDMC Coumaroyl triacetic acid synthase OS=Hydrangea macrophylla GN=ctas PE=2 SV=1
MATKSVAVEEMCKAQKAGGPATILAIGTAVPSNCYYQSEYPDFYFRVTKSDHLTDLKSKFKRMCERSSIKKRYMHLTEEILEENPNMCTFAAPSIDGRQDIVVKEIPKLAKEAASKAIKEWGQPKSNITHLVFCTTSGVDMPGCDYQLTRLLGLRPSIKRLMMYQQGCHAGGTGLRLAKDLAENNKGARVLVVCSEMTVINFRGPSEAHMDSLVGQSLFGDGASAVIVGSDPDLSTEHPLYQIMSASQIIVADSEGAIDGHLRQEGLTFHLRKDVPSLVSDNIENTLVEAFTPILMDSIDSIIDWNSIFWIAHPGGPAILNQVQAKVGLKEEKLRVSRHILSEYGNMSSACVFFIMDEMRKRSMEEGKGTTGEGLEWGVLFGFGPGFTVETIVLHSVPI
>sp|P13416|CHS1_SINAL Chalcone synthase 1 OS=Sinapis alba GN=CHS1 PE=2 SV=1
MVMGTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKDNPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDADISAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDDVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSVEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPV
>sp|P13417|CHS3_SINAL Chalcone synthase 3 OS=Sinapis alba GN=CHS3 PE=2 SV=1
MVMGTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKDNPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAAIIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDDVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSKEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPV
>sp|P17818|CHSY_MATIN Chalcone synthase OS=Matthiola incana GN=CHS PE=2 SV=1
MVMGATSLDEIRKAQRADGPAGILGIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFQRMCDKSMIRKRHMHLTEDFLKENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLEEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRKKSAQDGVATTGEGLEWGVLFGFGPGLTVETVVLRSVPL
>sp|O22652|CHSY_RAPSA Chalcone synthase OS=Raphanus sativus GN=CHS PE=2 SV=1
MVGTTSSLDEIRKAQRADGPAGILAIGTANPANHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDVSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGITFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDDVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSLDDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPV
>tr|B1N7K5|B1N7K5_RAPSA Chalcone synthase OS=Raphanus sativus PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGPRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQAPFSDGAAALIVGSDPDISAGEKPIFETVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P1|Q460P1_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460R0|Q460R0_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460R2|Q460R2_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWSVLFGFGPGLTVETVVLHSVPL
>tr|Q460R8|Q460R8_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACILFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460S8|Q460S8_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMLGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q5FBU5|Q5FBU5_ARATH Mutant protein of chalcone synthase OS=Arabidopsis thaliana GN=AT5G13930.1 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTNLPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460Q4|Q460Q4_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAEDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q5FBU6|Q5FBU6_ARATH Mutant protein of chalcone synthase OS=Arabidopsis thaliana GN=AT5G13930.1 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCNEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460Q3|Q460Q3_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEKFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMMSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDGAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460S2|Q460S2_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGTLAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460R3|Q460R3_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDKIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460T2|Q460T2_ARATH Chalcone synthase family protein OS=Arabidopsis thaliana GN=At5g13930 PE=2 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMMSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P7|Q460P7_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAATFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVATVVLHSVPL
>tr|Q460P3|Q460P3_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRYVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P6|Q460P6_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTALRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGRALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P4|Q460P4_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPQSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P8|Q460P8_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRANGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRRRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460Q0|Q460Q0_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFADGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P2|Q460P2_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQTEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRRRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFCGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P0|Q460P0_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAKNNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460P9|Q460P9_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVPAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460Q1|Q460Q1_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGAVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLFKDVPGLISKNIVKSLDEAFKPMGISDWNSPFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q460N8|Q460N8_ARAHG Chalcone synthase family protein OS=Arabidopsis halleri subsp. gemmifera GN=CHS5 PE=2 SV=1
MVMAAGASSLDEIRKAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSMIRKRHMHLTEDFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQAPFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAKDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|F1C7T4|F1C7T4_9BRAS Chalcone synthase OS=Parrya nudicaulis GN=CHS PE=2 SV=1
MVMGATSLDEIRKAQRADGPAGILGIGTANPANHVIQTEYPDYYFRITNSEHMTELKEKFQRMCDKSMIRKRHMHLTEEFLKENPNMCEYMAPSLDVRQDIVVVEVPKLGKEAAVRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLEEAFKPLGISDWNSLFWVAHPGGPAILDQVELKLGLKAEKMRATRHVLSEFGNMSSACVLFILDEMRKKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLRSVPL
>tr|F1C7T6|F1C7T6_9BRAS Chalcone synthase OS=Parrya nudicaulis GN=CHS PE=2 SV=1
MVMGATSLDEIRKAQRADGPAGILGIGTANPANHVIQTEYPDYYFRITNSEHMTELKEKFQRMCDKSMIRKRHMHLTEEFLKENPNMCEYMAPSLDVRQDIVVVEVPKLGKEAAVRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEKPIFEMVSAXQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLEEAFKPLGISDWNSLFWVAHPGGPAILDQVELKLGLKAEKMRATRHVLSEFGNMSSACVLFILDEMRKKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLRSVPL
>tr|B1N7K6|B1N7K6_BARVU Chalcone synthase OS=Barbarea vulgaris PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGPRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7J9|B1N7J9_ORYVI Chalcone synthase OS=Orychophragmus violaceus PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGPRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|Q84V00|Q84V00_BRAOL Chalcone synthase OS=Brassica oleracea GN=chs PE=2 SV=1
MGTPSSLSEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMIDLKEKFKRMCDKSGIRKRHMHLTEEFLEDNPNMCAYMAPSLDVRQDVVVVEVPKLGKEAAEKAIKEWGQPKSRITHLVFCTTSGVDMPGGDYQLTKLLGLCPSVKRLMMYQQGCFAGATVLRLAKDLAENNRGARVLVVCSEIIALFFRGPSDTHLDSLLGQALFSDGAAALVVGSDPDISVGEKPIFEMVSAAQTILPNSDGAINLQLREAGLTFHLLKHVPRLISKNIEKSLHEAFKPLGISDWNSLFWIAHPGGRAILDEVEKKLGLKAEKMRATRHVLSEYGNMTSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGYGPGLTVETVVLHSVPL
>tr|G9B9Y8|G9B9Y8_BRAOT Chalcone synthase OS=Brassica oleracea var. italica GN=CHS2 PE=2 SV=1
MGTPSSLGEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMIDLKEKFKRMCDKSGIRKRHMHLTEEFLKDNPNMCAYMAPSLDVRQDVVVVEVPKLGKEAAEKAIKEWGQPKSRITHLVFCTTSGVDMPGADYQLTKLLGLCPSVKRLMMYQQGCFAGATVLRLAKDLAENNRGARVLVVCSEIIALFFRGPSDTHLDSLLGQALFSDGAAALVVGSDPDTSVGEKPIFEMVSAAQTILPNSDGVINLQLREAGLTFHLLKHVPRLISENIEKSLHEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|J7EJA7|J7EJA7_BRAOT Chalcone synthase OS=Brassica oleracea var. italica PE=2 SV=1
MGTPSSLSEIRKAQRADGPAGILAIGTAYPANHVLQAEYPDYYFRITNSEHMLDLKEKFKRMCDKSGIRKRHMHLTEEFLEDNPNMCAYMAPSLDVRQDVVVVEVPKLGKEAAEKAIKEWGQPKSRITHLVFCTTSGVDMPGGDYQLTKLLGLCPSVKRLMMYQQGCFAGATVLRLAKDLAENNRGARVLVVCSEIIALFFRGPSDTHLDSLLGQALFSDGAAALVVGSDPDISVGEKPIFEMVSAAQTILPNSDGAINLQLREAGLTFHLLKHVPRLISKNIEKSLHEAFKPLGISDWNSLFWIAHPGGRAILDEVEKKLGLKAEKMRATRHVLSEYGNMTSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGYGPGLTVETVVLHSVPL
>tr|B1N7K4|B1N7K4_BRARA Chalcone synthase OS=Brassica rapa PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|D5LQ18|D5LQ18_BRARA Chalcone synthase 4 protein OS=Brassica rapa GN=CHS4 PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTISKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGERVGVGCLVRFRTSLTVETVVLHSVPL
>tr|B1N7K0|B1N7K0_BRARC Chalcone synthase OS=Brassica rapa var. parachinensis PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEK FKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIK EWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAK DLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDADTSAGEK PIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISD WNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSA EDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7J8|B1N7J8_BRAJU Chalcone synthase OS=Brassica juncea var. multiceps PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDISAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7F0|B1N7F0_BRARC Chalcone synthase OS=Brassica rapa subsp. chinensis GN=CHS2 PE=2 SV=1
MVMCTPSSLDEIGKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGIGDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7K3|B1N7K3_BRARC Chalcone synthase OS=Brassica rapa subsp. chinensis PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDISAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|A9P3A7|A9P3A7_BRARC Chalcone synthase OS=Brassica rapa subsp. chinensis PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7J7|B1N7J7_BRARC Chalcone synthase OS=Brassica rapa var. purpuraria PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7K7|B1N7K7_BRAJU Chalcone synthase OS=Brassica juncea PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDISAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|O81477|O81477_BRANA Chalcone synthase A2 OS=Brassica napus GN=CHSA2 PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMSPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGSTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDDVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSTEDGVGTTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|O81478|O81478_BRANA Chalcone synthase B1 OS=Brassica napus GN=CHSB1 PE=2 SV=1
MVMGPSSLDEIRKAQRADGPAGILAIGTANPANHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPNMCAYMAPSLDARQDLVVVEVPKLGKDAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLVSLVGQALFSDGAAALIVGSDPDVSAGEKPIFQMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDDVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSKEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|D2CFP6|D2CFP6_BRANA Chalcone synthase OS=Brassica napus GN=CHS PE=2 SV=1
MVMGPSSLDEIRKAQRADGPAGILAIGTANPANHVLQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPNMCAYMAPSLDARQDLVVVEVPKLGKDAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDISAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7K2|B1N7K2_BRAOC Chalcone synthase OS=Brassica oleracea var. capitata PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEIIAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|B1N7K1|B1N7K1_RORIN Chalcone synthase OS=Rorippa indica PE=2 SV=1
MVMCTPSSLDEIRKAQRADGPAGILAIGTANPANHVIQAEYPDYYFRITNSEHMTDLKEKFKRMCDKSTIRKRHMHLTEEFLKENPDMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGPRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSAGEKPIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLDEAFKPLGISDWNSLFWIAHPGGPAILDEVEKKLGLKAEKMRATRHVLSEYGNMSSACVLFILDEMRRKSAEDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL
>tr|G3FJ87|G3FJ87_9ASPA Chalcone synthase OS=Freesia hybrid cultivar PE=2 SV=1
MVNVEEIRKAQRAEGPAAILAIGTATPPNAIEQSEYPDYYFRVTNSEDKVELKEKFKRMCEKSMIKKRYLYLTEDILKENPNVCAYMATSLDARQDMVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSESHLDSLVGQALFGDGAAALIVGSDAIEGIERPIFEMVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGIISKNIEKSLEEAFKPLGITDYNSLFWIAHPGGPAILDQVEAKIGLKPEKLRATRHVLSEYGNMSSACVLFILEEMRKKSAEEKNGTTGEGLEWGVLFGFGPGLTVETVVLHSVEA
>tr|A9ZTK7|A9ZTK7_IRIHO Chalcone synthase OS=Iris hollandica GN=IhCHS2 PE=2 SV=1
MANIEEFRRAQRAEGPATVLAIGTATPSNVVYQSEYPDYYFRITNSEHLTDLKEKFKRMCDKSMIRKRYMHLDEEILKQNPDMCAYMAPSLDARQDIVVVEVPKLGKAAASAAIKEWGRPKSLITHVVFCTTSGVDMPGADYQLTKLLGLRPSVNRLMMYQQGCFAGGTVLRVAKDLAENNRGARVLVVCSEVTAVTFRGPSETHLDSLVGQALFGDGAAAMIIGSDPDVSVERPLFQLMYAQQTIVPDSQGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFGPLGISDWNSIFWVAHPGGPAILDAVEDKLGLEKAKMGATREVLKEYGNMSSACVIFILDEMRKRSAEGGKATYGEGLDMGVLFGFGPGLTVECVVLKGCPVAAPCVIHS
>tr|A9ZTK6|A9ZTK6_IRIHO Chalcone synthase OS=Iris hollandica GN=IhCHS3 PE=2 SV=1
MANIEEFRRAQRAEGPATVLAIGTATPSNVVYQSEYPDYYSASPTASTSPTSRPSSREWCEKSMISKRYMHLNEEILREQPNMCAYMAPSLDARQDIVVVEVPKLGKAAASAAIREWGRPKSLITHVVFCTTSGVDMPGADYQLAKLLGLRPSVNRLMMYQQGCFAGGTVLRVAKDLAENNRGARVLVVCSEITAVTFRGPSETHLDSLVGQALFGDGAAALIVGSDPDPSVERPLFQLVSAQQTIVPDSQGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLAEAFGPLGISDWNSIFWVAHPGGPAILDAVEDKLGLEKTKMGATREVLKEYGNMSSACVIFILDQMRKKSAEEGKATYGEGLDMGVLFGFGPGLTVECVVLKSCPVAAA
>tr|Q2ABX7|Q2ABX7_IRIHO Chalcone synthase OS=Iris hollandica GN=IhCHS1 PE=2 SV=1
MASIEEFRRAQRADGPAALLAIGTATPSNVVYQSEYPDYYFRITNSEHLTDLKEKFKRMCDKSMIRKRYMHLDEEILKQNPDMCAYMAPSLDARQDIVVVEVPKLGKAAASAAIKEWGRPKSLITHVVFCTTSGVDMPGADYQLTKLLGLRPSVNRLMMYQQGCFAGGTVLRVAKDLAENNRGARVLVVCSEVTAVTFRGPSETHLDSLVGQALFGDGAAAMIIGSDPDVSVERPLFQLMYAQQTIVPDSQGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFGPLGISDWNSIFWVAHPGGPAILDAVEDKLGLEKAKMGATREVLKEYGNMSSACVIFILDEMRKRSAEGGKATYGEGLDMGVLFGFGPGLTVECVVLKGCPVAAPCVIHS
>tr|Q2WFX2|Q2WFX2_IRIGE Chalcone synthase OS=Iris germanica GN=IgCHS1 PE=2 SV=1
MASVAEIRKAQRAEGPAAILAIGTANPPTAVAQSEYPDYYFRITNSEDKHELKEKFKRMCEKSMIKNRYMYLTEKILKENPSVCAYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLIFCTTSGVDMPGADYQLTRLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPSECHLDSLVGQALFGDGAAALIVGADPIENVERPLFEMVSAAQTILPDSEGAIDGHLREAGLTFHLLKDVPGIISKNIEKSLEEAFKPLGIADWNSLFWIAHPGGPAILDQVEAKIGLKPEKLRATRHVLSEFGNMSSACVLFILDEMRKRSVEEGNATTGEGLEWGVLFGFGPGLTVETVVLHSVAAA
>sp|A2ZEX7|CHS1_ORYSI Chalcone synthase 1 OS=Oryza sativa subsp. indica GN=CHS1 PE=2 SV=1
MAAAVTVEEVRRAQRAEGPATVLAIGTATPANCVYQADYPDYYFRITKSEHMVELKEKFKRMCDKSQIRKRYMHLTEEILQENPNMCAYMAPSLDARQDIVVVEVPKLGKAAAQKAIKEWGQPRSRITHLVFCTTSGVDMPGADYQLAKMLGLRPNVSRLMMYQQGCFAGGTVLRVAKDLAENNRGARVLAVCSEITAVTFRGPSESHLDSMVGQALFGDGAAAVIVGSDPDEAVERPLFQMVSASQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIERALGDAFTPLGISDWNSIFWVAHPGGPAILDQVEAKVGLDKERMRATRHVLSEYGNMSSACVLFILDEMRKRSAEDGHATTGEGMDWGVLFGFGPGLTVETVVLHSVPITAGAAA
>sp|Q2R3A1|CHS1_ORYSJ Chalcone synthase 1 OS=Oryza sativa subsp. japonica GN=CHS1 PE=2 SV=1
MAAAVTVEEVRRAQRAEGPATVLAIGTATPANCVYQADYPDYYFRITKSEHMVELKEKFKRMCDKSQIRKRYMHLTEEILQENPNMCAYMAPSLDARQDIVVVEVPKLGKAAAQKAIKEWGQPRSRITHLVFCTTSGVDMPGADYQLAKMLGLRPNVNRLMMYQQGCFAGGTVLRVAKDLAENNRGARVLAVCSEITAVTFRGPSESHLDSMVGQALFGDGAAAVIVGSDPDEAVERPLFQMVSASQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIERALGDAFTPLGISDWNSIFWVAHPGGPAILDQVEAKVGLDKERMRATRHVLSEYGNMSSACVLFILDEMRKRSAEDGHATTGEGMDWGVLFGFGPGLTVETVVLHSVPITAGAAA
>sp|Q96562|CHS2_HORVU Chalcone synthase 2 OS=Hordeum vulgare GN=CHS2 PE=2 SV=1
MAAVRLKEVRMAQRAEGLATVLAIGTAVPANCVYQATYPDYYFRVTKSEHLADLKEKFQRMCDKSMIRKRHMHLTEEILIKNPKICAHMETSLDARHAIALVEVPKLGQGAAEKAIKEWGQPLSKITHLVFCTTSGVDMPGADYQLTKLLGLSPTVKRLMMYQQGCFGGATVLRLAKDIAENNRGARVLVVCSEITAMAFRGPCKSHLDSLVGHALFGDGAAAAIIGADPDQLDEQPVFQLVSASQTILPESEGAIDGHLTEAGLTIHLLKDVPGLISENIEQALEDAFEPLGIHNWNSIFWIAHPGGPAILDRVEDRVGLDKKRMRASREVLSEYGNMSSASVLFVLDVMRKSSAKDGLATTGEGKDWGVLFGFGPGLTVETLVLHSVPVPVPTAASA
>tr|B5LXZ9|B5LXZ9_HORVU Chalcone synthase OS=Hordeum vulgare PE=2 SV=1
MAATMTVEEVRNAQRAEGPATVLAIGTATPANCVYQADYPDYYFKITKSDHMADLKEKFKRMCDKSQIRKRYMHLTEEILEENPNMCAYMAPSLDARQDIVVVEVPKLGKAAAQKAIKEWGQPRSKITHLVFCTTSGVDMPGADYQLTKMLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPHESHLDSLVGQALFGDGAAAVIIGADPDVSVERPLFQLVSASQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIERALEDAFKPLGIDDWNSVFWIAHPGGPAILDMVEAKVNLNKERMRATRHVLSEYGNMSSACVLFIMDEMRKRSAEDGHTTTGEGMDWGVLFGFGPGLTVETVVLHSVPITA
>sp|P53415|CHS2_SECCE Chalcone synthase 2 OS=Secale cereale GN=CHS2 PE=2 SV=1
MAATMTVEEVRKAQRAEGPATVLAIGTATPANCVYQADYPDYYFKITKSDHMADLKEKFKRMCDKSQIRKRYMHLTEEILQDNPNMCAYMAPSLDARQDIVVVEVPKLGKAAAQKAIKEWGQPRSKITHLVFCTTSGVDMPGADYQLTKMLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPHESHLDSLVGQALFGDGAAAVIIGADPDESIERPLFQLVSASQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIERALEDAFKPLGIDDWNSVFWIAHPGGPAILDMVEAKVNLNKERMRATRHVLSEYGNMSSACVLFIMDEMRKRSAEDGHTTTGEGMDWGVLFGFGPGLTVETVVLHSVPVTA
>tr|Q4QWZ6|Q4QWZ6_MAIZE Chalcone synthase OS=Zea mays GN=c2 PE=2 SV=1
MAGATVTVEEVRKAQRATGPATVLAIGTATPANCVYQADYPDYYFRITKSEHLTDLKEKFKRMCDKSMIRKRYMHLTEEFLAENPSMCAYMAPSLDARQDVVVVEVPKLGKAAAQKAIKEWGQPKSRITHLVFCTTSGVDMPGADYQLTKALGLRPSVNRLMMYQQGCFAGGTVLRVAKDLAENNRGARVLVVCSEITAVTFRGPSESHLDSLVGQALFGDGAAAVVVGADPDDRVERPLFQLVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIGRALDDAFKPLGISDWNSIFWVAHPGGPAILDQVEAKVGLDKARMRATRHVLSEYGNMSSACVLFILDEMRKRSAEDGQATTGEGLDWGVLFGFGPGLTVETVVLHSVPITTGAATA
>tr|Q8VWQ7|Q8VWQ7_SORBI Chalcone synthase 8 OS=Sorghum bicolor GN=Sb07g004700 PE=2 SV=1
MTTGKVTLEAVRKAQRAEGPATVLAIGTATPANCVYQADYPDYYFRVTKSEHLTDLKEKFKRICHKSMIRKRYMHLTEDILEENPNMSSYWAPSLDARQDILIQEIPKLGAEAAEKALKEWGQPRSRITHLVFCTTSGVDMPGADYQLIKLLGLCPSVNRAMMYHQGCFAGGMVLRLAKDLAENNRGARVLIVCSEITVVTFRGPSESHLDSLVGQALFGDGAAAVIVGADPSEPAERPLFHLVSASQTILPDSEGAIEGHLREVGLTFHLQDRVPQLISMNIERLLEDAFAPLGISDWNSIFWVAHPGGPAILNMVEAKVGLDKARMCATRHILAEYGNMSSVCVLFILDEMRNRSAKDGHTTTGEGMEWGVLFGFGPGLTVETIVLHSVPITTVAA
>tr|Q6WGP9|Q6WGP9_WHEAT Chalcone synthase OS=Triticum aestivum GN=CHS PE=2 SV=1
MAATMTVEEVRKAQRAEGPATVLAIGTATPANCVYQADYPDYYFKITKSDHMADLKEKFKRMCDKSQIRKRYMHLTEEILQDNPNMCAYMAPSLDARQDIVVVEVPKLGKAAAQKAIKEWGQPRSKITHLVFCTTSGVDMPGADYQLTKMLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVVCSEITAVTFRGPHESHLDSLVGQALFGDGAAAVIIGADPDESIERPLFQLVSASQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIERALEDAFKPLGIDDWNSVFWIAHPGGPAILDMVEAKVNLNKERMRATRHVLSEYGNMSSACVLFIMDEMRKRSAEDGHSTTGEGMDWGVLFGFGPGLTVETVVLHSVPVTA
>tr|Q6WGP8|Q6WGP8_WHEAT Chalcone synthase OS=Triticum aestivum GN=CHS1 PE=2 SV=1
MAATMTVEEVRKAQRAEGPATVLAIGTATPANCVYQADYPDYYFKITKSDHMADLKEKFKRMCDKSQIRKRYMHLTEEILQDNPNMCAYMAPSLDARQDIVVVEVPKLGKAAAQKAIKEWGQPRSKITHLVFCTTSGVDMPGADYQLTKMLGLCPSVKRLMMYQQGCFAGGTVLRLAKDLVENNRGARVLVVCSEITAVTFRGPHESHLDSLVGQALFGDGAAAVIIGADPDESIERPLFQLVSASQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIERALEDAFKPLGINDWNSVFWIAHPGGPAILDMVEAKVNLNKERMRATRHVLSEYGNMSSACVLFIMDEMRKRSAEDGHTTTGEGMDWGVLFGFGPGLTVETVVLHSVPVTA
>tr|D6PV09|D6PV09_SIRGR Chalcone synthase OS=Siraitia grosvenorii PE=2 SV=1
MASVEQIRKAQKADGPATVLAIGTATPPNSVLQADYPDYYFRITNSEHMTELKEKFRRMCDKSMIRKRFMYLTEEILTENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAAKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLVKLLGLRPSVKRYMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSHAHLDSLVGQALFGDGAAAVIIGSDPDLDIERPLYELVWTGTTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLKEAFTPLGISDWNSIFWIAHPGGPAILDQVEAKLGLKEEKMRATREILSEYGNMSSACVLFIMDQMRKNSMEEGKTTTGEGLEWGVLFGFGPGLTVETVVLHSVNVKETTVKAAN
>tr|M4PZZ8|M4PZZ8_RHUCH Chalcone synthase OS=Rhus chinensis PE=2 SV=1
MVSVDEVRKAQRAEGPATVMAIGTATPPNCVDQSTYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPAVCEYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRYMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAAVIVGSDPVPGVEKPMFELVSTAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGISDWNSLFWIAHPGGPAILDQVEIKLGLKEEKLRATRHVLSEYGNMSSACVLFILDEMRKKSTENGLKTTGEGLEWGVLFGFGPGLTVETVVLHSIATT
>tr|Q2WBN0|Q2WBN0_9LAMI Chalcone synthase OS=Misopates orontium GN=chs PE=2 SV=1
MVTVEEVRRAQRAEGPATVLAIGTATPANCVDQSTYPDYYFRITNSEHMTDLKEKFKRMCEKSMIKKRYMHLTEEILKENPAMCEYMAPSLDARQDIVVVEVPRLGKEAAQKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRMAKDLAENNAGARVLVVCSEITAVTFRGPADTHLDSLVGQALFGDGAAAVIVGSDPVVGVERPLFQLVTAAQTLLPDSHGAIDGHLREVGLTFHLLKDVPGLISKHIEKSLKEAFDPLGISDWNSVFWIAHPGGPAILDQVEEKLGLKPEKLRSTRHVLSEYGNMSSACVLFILDEMRKSSTKEGMSTTGEGFDWGVLFGFGPGLTVETVVLHSVPLN
>tr|Q8W123|Q8W123_HYPPE Chalcone synthase OS=Hypericum perforatum PE=2 SV=1
MVTVEEVRKAQRAEGPATVMAIGTAVPPNCVDQATYPDYYFRITNSEHKAELKEKFQRMCDKSQIKKRYMYLNEEILKENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAASIIIGSDPIPEVEKPLFELVSASQTILPDSEGAIDGHIREVGLTFHLLKDVPGLISKNVEKSLTEAFKPLGISDWNSLFWIAHPGGPAILDQVEEKLSLKPEKLRATRHVLSEYGNMSSACVLFILDEMRRKSKEDGLKTTGEGIEWGVLFGFGPGLTVETVVLHSVAY
>tr|K4HPM7|K4HPM7_9ROSI Chalcone synthase OS=Hypericum sampsonii PE=2 SV=1
MVTVEEVRKAQRAEGPATVMAIGTAVPPNCVDQATYPDYYFRITNSEHKAELKEKFQRMCDKSQIKKRYMYLNEEVLKENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAAIIIGSDPIPEVEKPLFELVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNVEKSLTEAFKPLGISDWNSLFWIAHPGGPAILDQVEAKLSLKPEKLRATRHVLSEYGNMSSACVLFILDEMRRKSKEDGLKTTGEGIEWGVLFGFGPGLTVETVVLHSLAIN
>tr|B9UZ48|B9UZ48_GARMA Chalcone synthase OS=Garcinia mangostana GN=CHS PE=2 SV=1
MAPTVEEVRNAQRAQGPATVLAIGTATPSNCVLQAEYPDYYFRITNSEHKTELKEKFRRMCEKSMIKKRYMHLTEEILKENPKMCDYWSPSLDARQDIVVVEIPKLGKEAAVKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPHVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVICSEITAVTFRGPSDTHLDSLVGQALFGDGAAAIIVGSDPDPAIERPLFQIVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFTPIGISDWNSLFWIAHPGGPAILDQVEVKLGLKEEKLRATRHVLSEFGNMSSACVLFILDEMRKKALEEGKPTTGEGLDWGVLFGFGPGLTVETVVLHSVPTETRA
>tr|I6TGM5|I6TGM5_ACEPM Chalcone synthase OS=Acer palmatum PE=2 SV=1
MVTVDEVRKAQRAQGPATILAIGTATPPNCVDQSEYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMYLTEEILKENPNVCAYEAPSLDARQDMVVVEIPKLGKEAATKAIKEWGQPKSKITHLVFCSTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNTGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAIIIGSDPQPGIEKPMYELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFKPLGISDWNSIFWIAYPGGPAILDQVEAKLGLKPEKMRATRHVLSEFGNMSSACVLFILDEMRKKSAEDGLRTTGEGLEWGVLFGFGPGLTVETVILHSVSAA
>sp|P51075|CHSY_BETPN Chalcone synthase OS=Betula pendula GN=CHS PE=2 SV=2
MASVEEIRKAQRAHGPATVLAIGTATPSNCITQADYPDYYFRITKSDHMTELKEKFKRMCDKSMIKKRYMYLNEEILNENPNMCAYMAPSLDARQTIVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAAVIVGADPDTSVERPLFELISAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGIISKNIEKSLAEAFAPLGISDWNSLFWIAHPGGPAILDQVESKLGLKEEKLRATRHVLSEYGNMSSACVLFILDEMRRNSLEGGKVTTGEGLEWGVLFGFGPGLTVETVVLHSVPVPVEASH
>sp|P48390|CHS1_GERHY Chalcone synthase 1 OS=Gerbera hybrida GN=CHS1 PE=2 SV=1
MASSVDMKAIRDAQRAEGPATILAIGTATPANCVYQADYPDYYFRITKSEHMVDLKEKFKRMCDKSMIRKRYMHITEEYLKQNPNMCAYMAPSLDVRQDLVVVEVPKLGKEAAMKAIKEWGHPKSKITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGAAAVIVGSDPDLTTERPLFEMVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIEKALTTAFSPLGINDWNSIFWIAHPGGPAILDQVELKLGLKEEKLRATRHVLSEYGNMSSACVLFIIDEMRKKSSENGAGTTGEGLEWGVLFGFGPGLTVETVVLHSVPTTVTVAV
>tr|B7ZF46|B7ZF46_GERHY Chalcone synthase OS=Gerbera hybrida GN=gchs4 PE=2 SV=1
MVNIEEFRKAQRAEGPATIMAIGTATPSNCVFQDTYPDYYFRVTKSEHKTELKEKFKRMCDKSMIKKRYMYLTEEILEEKPNVCAYMAPSLNERQDIVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPDETHLDSLVGQALFGDGAAAIIVGSDPLLGQEKPLFEMVYAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKHIDKSLVEAFQPLGITDWNSLFWVAHPGGPAILDQVEEKLGLEPDKLRATRHVLSEYGNMSSACVLFILNEMRHSSATDGLKTTGEGLEWGVLFGFGPGLTVETVVLHSVPI
>sp|P48385|CHSY_CALCH Chalcone synthase OS=Callistephus chinensis GN=CHS PE=2 SV=2
MASTIDIAAIREAQRRQGPATILAIGTATPSNCVYQADYPDYYFRITKSEHMVDLKEKFKRMCDKSMIRKRYMHLTEEYLKENPSLCEYMAPSLDARQDVVVVEVPKLGKEAATKAIKEWGQPKSKITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGAAAVIVGADPDLTTERPLFEMISAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIEKALTQAFSPLGITDWNSIFWIAHPGGPAILDQVELKLGLKEEKMRATRHVLSEYGNMSSACVLFIIDEMRKKSAEDGAATTGEGLDWGVLFGFGPGLTVETVVLHSLPTTMAIAT
>tr|D9N563|D9N563_DAHPI Chalcone synthase 1 OS=Dahlia pinnata GN=CHS1 PE=2 SV=1
MVSIQEFRNAQRADGPAAILAIGTATPPNCVLQSEYPDYYFRVTKSEHKKDLKEKFTRMCEKSMIRKRYMYLTEEILKEKPNICAYMAPSLDERQDIVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRSSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPDNTHLDSLVGQALFGDGAAAIIVGSDPLPDVEKPLFDIISAGQTILPDSGGAIDGHLREVGLTFHLLKDVPGLISKHIETSLVDAFQPLGINDWNSLFWIAHPGGPAILDQVEEKLALTPDKLQASRHVLSEYGNMSSACVLFILNEMRHSSATDGFNTTGEGLEWGVLFGFGPGLTVETVVLHSVSI
>tr|D9N565|D9N565_DAHPI Chalcone synthase type 2 OS=Dahlia pinnata GN=CHS1 PE=2 SV=1
MVSIQEFRNAQRADGPAAILAIGTATPPNCVLQSEYPDYYFRVTKSEHKKDLKEKFTRMCEKSMIRKRYMYLTEEILKEKPNICAYMAPSLDERQDIVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRSSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPDDTHLDSLVGQALFGDGAAAIIVGSDPLPDVEKPLFEIISAGQTILPDSGGAIDGHLREVGLTFHLLKDVPGLISKHIETSLVDAFQPLGINDWNSLFIAHPGGPAILDQVEEKLALTPDKLRASRHVLSEYGNMSSACVLFILNEMRHSSATDGFNTTGEGLEWGVLFGFGPGLTVETVVLHSVSI
>tr|D9N566|D9N566_DAHPI Chalcone synthase 2 OS=Dahlia pinnata GN=CHS2 PE=2 SV=1
MASSVDIAAFREAQRAEGPATILAIGTATPPNCLYQADYPDYYFRITNSEHMVELKEKFKRMCDKSMIRKRYMHLTEEYLKEHPNICEYMAPSLDARQDVVVVEVPKLGKDAAVKAIKEWGKPKSQITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAALIVGSDPDLTTERPLFQMISAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLIAKNIEKALVQAFSPLGISDWNSIFWIAHPGGPAILDQVESKLGLKEEKMRATRHVLGEYGNMSSACVLFILDEMRKKSAEDGVGTTGEGLDWGVLFGFGPGLTVETVVLHSVPTTMPISP
>tr|E1CPW6|E1CPW6_DAHPI Chalcone synthase type 2 OS=Dahlia pinnata GN=CHS2 PE=2 SV=1
MASSVDIAAFREAQRAEGPATILAIGTATPPNCLYQADYPDYYFRITNSEHMVELKEKFKRMCDKSMIRKRYMHLTEEYLKEHPNICEYMAPSLDARQDVVVVEVPKLGKDAAVKAIKEWGKPKSQITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAALIVGSDPDLATERPLFQMISAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLIAKNIEKALVQAFSPLGISDWNSIFWIAHPGGPAILDQVESKLGLKEEKMRATRHVLGEYGNMSSACVLFILDEMRKKSAEDCVGTTGEGLDWGVLFGFGPGLTVETVVLHSVPTTMPISP
>tr|D9N564|D9N564_DAHPI Chalcone synthase type 3 OS=Dahlia pinnata GN=CHS1 PE=2 SV=1
MVSIQEFRNAQRADGPAAILAIGTATPPNCVLQSEYPDYYFRVTKSEHKKDLKEKFTRMCEKSMIRKRYMYLTEEILKEKPNICAYMAPSLDERQDIVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRFSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPDDTHLDSLVGQALFGDGAAAIIVGSDPLPDVEKPLFEIISAGQTILPDSGGAIDGHLREVGLTFHLLKDVPGLISKHIETSLVDAFQPLGINDWNSLFWIAHPGGPAILDQVEEKLALTPDKLQASRHVLSEYGNMSSACVLFILNEMRHSSATDGFNTTGEGLEWGVLFGFGPGLTVETVVLHSVSI
>tr|D9N562|D9N562_DAHPI Chalcone synthase type 1 OS=Dahlia pinnata GN=CHS1 PE=2 SV=1
MVSIQEFRNAQRADGPAAILAIGTATPPNCVLQSEYPDYYFRVTKSEHKKDLKEKFTRMCEKSMIRKRYMYLTEEILKDKPNICAYMAPSLDERQDIVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRSSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPDNTHLDSLVGQALFGDGAAAIIVGSDPLPDVEKPLFEIISAGQTILPDSGGAIDGHLREVGLTFHLLKDVPGLISKHIETSLVDAFQPLGINDWNSLFWIAHPGGPAILDQVEEKLALTPDKLQASRHVLSEYGNMSSACVLFILNEMRHSSATDGFNTTGEGLEWGVLFGFGPGLTVETVVLHSVSI
>tr|E0D860|E0D860_DAHPI Chalcone synthase type 1 OS=Dahlia pinnata GN=CHS1 PE=2 SV=1
MVSIQEFRNAQRADGPAAILAIGTATPPNCVLQSEYPDYYFRVTKSEHKKDLKEKFTRMCEKSMIRKRYTYLTEEILKEKPNICAYMAPSLDERQDIVVVEVPKLGKEAATRAIKEWGQPKSNITHLVFCTTSGVDMPGADYQLTKLLGLRSSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPDNTHLDSLVGQALFGDGAAAIIVGSDPLPDVEKPLFEIISAGQTILPDSGGAIDGHLREVGLTFHLLKDVPGLISKHIETSLVDAFQPLGINDWNSLFWIAHPGGPAILDQVEEKLALTPDKLQASRHVLSEYGNMSSACVLFILNEMRHSSATDGFNTTGEGLEWGVLFGFGPGLTVETVVLHSVSI
>tr|A1XEV4|A1XEV4_SAUME Chalcone synthase OS=Saussurea medusa GN=CHS PE=2 SV=1
MVTVEEVRKATRAEGPATIMAIGTATPPNCVLQSAYPDYYFRVTKSEHKKELKEKFRRMCDKSMIKKRYMHLTEEILQEKPNICAYMAPSLDERQDIVVVEVPKLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPNETHLDSLVGQALFGDGAAAIIVGSDPLLGVEKPLFEIVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKHIEKSLLEAFRPLGIFDWNSLFWIAHPGGPAILDQVEEKLSLKPDKLRATRHVLSEYGNMSSACVLFILNEMRHASATDGFNTTGEGLEWGVLFGFGPGLTVETLVLHSVSI
>tr|E0D6R7|E0D6R7_9ASTR Chalcone synthase OS=Gynura bicolor GN=GbCHS PE=2 SV=1
MASSIDIEKIREAQRAQGPATILAIGTATPNNCVYQADYPDYYFRITKSEHMVDLKEKFKRMCDKSMIRKRYMHLTEEYLKENQNLCEYMAPSLDARQDVVVVEVPKLGKEASTKAIKEWGQPKSKITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGAAAVIVGSDPDLTIERPLFEMISAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIEKALTQAFTPLGITDWNSLFWIAHPGGPAILDQVELKLGLKEEKMRATRHVLSEYGNMSSACVLFIIDEMRKKSAEDGAATTGEGLDWGVLFGFGPGLTVETVVLHSLPTTMHVAP
>tr|Q19LA8|Q19LA8_CHRMO Chalcone synthase OS=Chrysanthemum morifolium GN=CHS PE=2 SV=1
MASLTDIAAIREAQRAQGPATILAIGTATPANCVYQADYPDYYFRITKSEHMVDLKEKFKRMCDKSMIRKRYMHLTEEYLKENPSLCEYMAPSLDARQDVVVVEVPKLGKEAATKAIKEWGQPKSKITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKDARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGAAAVIVGSDPDLTKERPLFEMISAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIEKALTQAFSPLGISDWNSIFWIAHPGGPAILDQVELKLGLKEEKMRATRHVLSEYGNMSSACVLFIIDEMRKKSAEEGAATTGEGLDWGVLFGFGPGLTVETVVLHSLPTTISVA
>tr|A8QKF1|A8QKF1_RUDHI Chalcone synthase OS=Rudbeckia hirta PE=2 SV=1
MGSSIDIAAIREAQRLRSGPILSIGTATPSHCVYQADYPFYYFRITSEHMVDLKEKFKRMCDKSMIRKRYMHLTEEYLKENPNICEYMAPSLDARQDVVVVEVPKLGIEAAVKAIKEWGKPKSQITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGAAAVIVGSDPDLTTERPLFEMVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNIEKALTQAFSPLGITDWNSIFWIAHPGGPAILDQVELKLGLKEEKMRATRHVLSEFGNMSSACVLFILDEMRKKSAEEGAATTGEGLDWGVLFGFGPGLTVETVVLHSVPTTMMHAPQI
>sp|Q9ZRS4|CHSY_CATRO Chalcone synthase OS=Catharanthus roseus GN=CHS PE=2 SV=1
MVNVEEIRNAQRAQGPATVLAIGTSTPSNCVDQSTYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMHLTEEILQENPNICAYMAPSLDARQNIVVVEVPKLGKEAAQKAIKEWGQSKSKITHLVFCTTSGVDMPGADYQLTKLLGLRSSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSESHLDSLVGQALFGDGAAAIIVGSDPIPEIERPLFELVSAAQTLLPDSHGAIDGHLREVGLTFHLLKDVPGLISKNIGKALDEAFQPLGISDWNSIFWIAHPGGPAILDQVEEKLGLKPEKLRATRHVLSEYGNMSSACVLFILDEMRKASARDGLSTTGEGLEWGVLFGFGPGLTVETVVLHSVNV
>sp|Q9ZU06|CHSY_PERAE Chalcone synthase OS=Persea americana GN=CHS PE=2 SV=1
MVNVEAIRKVQRAEGPATIMAIGTSTPPNAVDQSEYPDYYYFGSPTASTRPSSRRSFKRMCEKSMIKKRYMYLTETYWKRIQMFVPTWLLPLKARQDMVVVEVPKLGKEVQPKAIKGMGQPKSKINPLVFCTTSGVDMPGADYQLTKVFGLPPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAALIVGADPVPGVENPMFELVSAGQTILPDSDGAIDGHLREVGLTFHLLKVVPGLISKNIEKSLVEAFEPLGISDWNSLFWIAHPGGPAILDPGGDQTRPEARESCGNQACFSVSMATCQVFVCSSFSTRCEGSPKEEGLKTTGEGIEWGVLFGFGPGLTVETVVLHSLPTH
>tr|Q42865|Q42865_9ROSI Naringenin-chalcone synthase OS=Juglans nigra x Juglans regia GN=CHS2 PE=2 SV=1
MVTVEDVRRAQRAEGPATVMAIGTATPPNCVDQSAYPDYYFRITNSEHKTELKEKFKRMCGKSMIKKRYMHLTEEILKENPNVCAYMASSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAALIVGADPVPGVEKPLFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGITDWNSLFWIAHPGGPAILDQVESKLELKPEKLRATRHVLSEYGNMSSACVLFILDEMRKKSAEDRLKTTGEGLEWGVLFGFGSGLTVETVVLHSVSA
>tr|Q42864|Q42864_9ROSI Naringenin-chalcone synthase OS=Juglans nigra x Juglans regia GN=CHS1 PE=2 SV=1
MVTVEDVRRAQRAEGPATVMAIGTATPPNCVDQSAYPDYYFRITNSEHKTELKEKFKRMCEKSMIKKRYMHLTEEILKENPNVCAYMASSLDARQDMVVVEVPKLGKEAATKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAALIVGADPVPGVEKPLFELVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLVEAFQPLGITDWNSLFWIAHPGGPAILDQVESKLELKPEKLRATRHVLSEYGNMSSACVLFILDEMRKKSAEDRLKTTGEGLEWGVLFGFGPGLTVETVVLHSVSA
>tr|Q7XB37|Q7XB37_MAZPU Chalcone synthase OS=Mazus pumilus GN=CHS PE=2 SV=1
MTPTVEEIRRAQRAEGPATVLAIGTATPSNCVDQSTYPDYYFRITNSEHMTDLKEKFKRMCEKSYIKKRYMHLTEEILKENPNMCAYMAPSLDARQDIVVVEIPKLGKEAAQKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRMAKDLAENNAGARVLVVCSEITAVTFRGPSDSHLDSLVGQALFGDGAAAVIVGSDPIVGVERPLFQIVSAAQTLLPDSHGAIDGHLREVGLTFHLLKDVPGLISKHIEKSLKEAFDPLGISDWNSIFWIAHPGGPAILDQVESVLALKPEKMRATRQVLSDYGNMSSACVLFILDEMRKASAKEGMGSTGEGLDWGVLFGFGPGLTVETVVLHSVPFMN
>tr|Q9FS37|Q9FS37_9LAMI Chalcone synthase OS=Torenia hybrid cultivar PE=2 SV=1
MVTVEEIRRAQRAEGPATILAIGTSTPANCVDQSTYPDYYFRITNSEHMTELKEKFKRMCEKSMIKKRYMHLTEDYLKENPNVCAYMAPSLDARQDIVVVEVPKLGKEAAQKAIKEWGQPKSKITHLIFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRMAKDLAENNAGARVLVVCSEITAVTFRGPSESHLDSLVGQALFGDGAAAVIVGSDPMSVERPLFQMVSAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGIISKNIEKSLKEAFDPLGISDWNSIFWNAHPGGPAILDQVEEKLGLKPDKLRATRTVLSEYGNMSSACVLFILDEMRKASAKEGLGSTGEGLDWGVLFGFGPGLTVETVVLHSTPN
>tr|Q76EK7|Q76EK7_9LAMI Chalcone synthase OS=Torenia hybrid cultivar GN=CHS PE=2 SV=1
MVIVEAIRRAQRAEGAATILAIGTSTPQNCVDQSTYPDYYFRITNSEHMTELKDKFTRMCEKSMIKKRYMHLTEDYLKQNPNICAYMEPSLDARQDIVVVEVPKLGKEAAQKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRMAKDLAENNAGARVLVVCSEITAVTFRGPSDAHLDSLVGQALFGDGAAAVIVGSDPTDVERPLFQLVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLKEAFDPLGIDDWNSIFWIAHPGGPAILDQVEAKLALKPDKLRATRHVLSEYGNMSSACVLFILDEMRKASAKEGTSTTGEGLDWGVLFGFGPGLTVETVVLHSVPLPNN
>tr|Q84L24|Q84L24_ANTAD Putative chalcone synthase OS=Anthurium andraeanum GN=CHS1 PE=2 SV=1
MVSTSLEAIRKAQRADGPATILAIGTAVPPNAVDQSTYPDYYFRITNSEHQVELKEKFRRMCEKSMIKKRHMYLTEEILKENPSMCAYMAPSLDARQDMVVVEVPRLGKEAATRAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLAKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAENNRGARVLVICSEVTAVTFRGPSESHLDSLVGQALFGDGASALVVGADPVEGVERPIFQVVSAAQTILPDSHGAIDGHLREVGLTFHLLKDVPGLISKNIDKSLVEAFEPLGISDWNSLFWIAHPGGPAILDQVEDKLRLQSEKLGATRRVLSEYGNMSSACVHFILDEMRKRSAEEGRGTTGEGLEWGVLFGFGPGLTVETLVLHSVAI
>sp|O04111|CHSY_PERFR Chalcone synthase OS=Perilla frutescens GN=CHS PE=2 SV=1
MVTVEDIRRAQRAEGPATVMAIGTATPENCVDQSTYPDYYFRITNSEHRTDLKEKFKRMCEKSMIRKRYMHLTEEFLKENPNMTAYMAPSLDARQDIVVVEVPKLGKEAAQKAIKEWGQPKSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRMAKDLAENNAGARVLVVCSEITAVTFRGPSESHLDSLVGQALFGDGAAAVIVGSDPVVGVERPLFQLVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLKEAFGPLGISDWNSVFWIAHPGGPAILDQVEAKLGLKPEKLRSTRHVLGEYGNMSSACVLFILDEMRKSSAKEGMSSTGEGLDWGVLFGFGPGLTVETVVLHSVPINN
>tr|A5A369|A5A369_SOLSC Chalcone synthase OS=Solenostemon scutellarioides PE=2 SV=1
MVTVEDIRRAQRAEGPATVLAIGTSTPSNCVDQSTYPDYYFRITNSEHKTDLKEKFKRMC
EKSMIRKRYMHLTEEFLKENPNMTAYMAPSLDARQDIVVVEVPKLGKEAAQKAIKEWGQP
KSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRFMMYQQGCFAGGTVLRMAKDLAEN
NAGARVLVVCSEITAVTFRGPSDSHLDSLVGQALFGDGAAAVIVGSDPVVGVERPLFQLV
SAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLKEAFGPLGITDWNSVFW
IAHPGGPAILDQVEAKLRLTPEKLRSTRHVLSEYGNMSSACVLFILDEMRKTSAKEGLNS
TGEGLDWGVLFGFGPGLTVETVVLHSVPLN