年を取ると、人は新しいことから順に忘れていくという。朝食後に血糖降下剤を飲んだかどうかは記憶にないくせに、40年以上前、K君が私が食べたかった大きい方のおにぎりを食べてしまったことなどは、まだ鮮明に憶えている。それは横に置いて、私が少しばかり遺伝子を扱い始めたのは55歳を過ぎてからである。この頃には様々な研究用のキットが販売され始めており、PCRで増幅された遺伝子断片の配列決定などは学部の学生でも容易にできるようになっていた。
研究の成りゆきから、自然界から分離した微生物の種を決める必要があった。微生物学の素養に乏しかった私にとって、従来からあった伝統的な分類手法は難し過ぎた。おのずとPCR法でリボソームタンパク質をコードする遺伝子の一部を増幅してその塩基配列を求め、この塩基配列をデータベースに登録されている配列と比較することにより種を決める(推定する)方法に流れたのは、仕方がなかったといえるだろう。このやり方は確かに簡単である。しかし、選択培地で分離した微生物を培養した後、顕微鏡で微生物の姿を見ることもなく殺し、DNAを取れば後はマニュアル通りにことが進むという研究スタイルには少なからぬ違和感を感じていた。研究室に良い顕微鏡がなかったことが一つの原因ではあったが、そうしたやり方では微生物に対して生き物としての共感を感じる場面が少ないのである。人間、共感を持ってやったことはなかなか忘れないが、違和感を感じながらこなした作業はすぐに忘れるものである。まして、学生さんに指示してやってもらっただけで自ら手を動かしていないとなれば、忘却のスピードはすばらしく速い。いや、一度は自ら手を下してやってみたのである。ところがピペットチップの先端が、マクロチューブの中に入っているかどうかが分かりにくかった。業者の人を捕まえてチップかチューブの色にコントラストをつけて見えやすくしたらと提案したのだが、「先生、その年でピペッティングする人なんかいませんよ。」と軽く却下されてしまった。
とはいえ、この際に使った遺伝子のデータベースを用いて忘れかけた記憶を辿りながら、植物体内でスチルベノイドとフラボノイドの分岐点で実際に機能している2種の酵素、カルコンシンターゼとスチルベンシンターゼのアミノ酸配列から、この2種の酵素の分かれた時代を推定しようと考えたわけである。データベースから酵素のアミノ酸配列を収集してこれを比較するという、慣れた人にとっては何でもない作業が、私にはとんでもない難しい作業になってしまっていた。当時、NCBI、DDBJ、GenBank 、UniProt、DADなどのデータベースを、ほとんど違和感なく使っていたというのに、いざ使おうとしたらどこに何があったか記憶が希薄になっていただけでなく、加齢に伴う一時記憶容量の著しい低下に改めて気付いてしまったのである。とはいえ、アブシジン酸について総合的に理解したいというライフワークを完結させるためには通らざるを得ない道である。そう考えて無謀な解析を始めてしまった。
さて、専門の人々には当たり前のことが、専門外の人にはとても難しく感じる例には事欠かない。私がたどたどしく行った初歩的な解析は、逆に一般の人に分かりやすいかもしれないと考え、少し冗長気味に書くことにする。
スチルベンシンターゼについて
ここでは、ルヌラリン酸とフラボノイドの分岐点で作用するカルコンシンターゼとスチルベンシンターゼのアミノ酸配列情報を基に、両酵素が分かれた時期を推定するのが目的である。この二つの酵素は別名で書かれているとはいえ、大きく見ればchalcone/stilbene synthases familyという同じ族に分類される酵素である。UniProtでChalcone synthaseをキーワードとして検索すると3,512 件がヒットするしstilbene synthaseとして検索すると3,382件の結果が表示される。(2011/11/07)前節で述べたゼニゴケのスチルベンシンターゼを比較する対象にすることをを考えると、この酵素のアミノ酸残基数392が一つの基準になるだろう。そこで、アミノ酸残基数を389から395 (392±3) の範囲に制限すると663件に絞ることができた。しかし、これらの結果はまだ玉石混淆で、酵素として発現している配列、酵素タンパクとしては確認されていないが転写レベルで確認されたもの、核酸の塩基配列から予想されただけのものなど多くの配列の混合体であり、そのまま利用するには余りにもノイズが多すぎる。つまり、この集団の中から解析に必要な配列を選ばなければならない。
どのような解析をするにしても、考える基礎を何処に置くのかを明確にしておかないと、考えが上滑りしてしまう。膨大なデータ群を対象とする場合は、いっそう注意する必要がある。では、スチルベンシンターゼ(stilbene synthase)、アミノ酸残基数389-395としての検索結果に、どのような条件を付け加えて数を絞るべきか。まず、タンパク質レベルでの発現が確認されているスチルベンシンターゼを使おうと考えて、この条件を加えて検索を行うと27件に減少する。ところが、この集団の中には、微生物由来のスチルベンシンターゼ、酵素としての反応メカニズムは同じで酵素としての起源は同一かもしれないにしても、現在の作用物質が違うクルクミン合成酵素やベンゾフェノン合成酵素、α-ピロン合成酵素などのデータも含まれるため、それらを除くと5つのデータしか残らない。これでは解析に使用するにはあまりにも少なすぎる。
少し条件をゆるめないといけないのだが、いわゆる核酸の塩基配列から相同なタンパク質であるとして類推されているだけのデータは使いたくない。生物の遺伝子上には、同じ機能を持つ二つ以上の遺伝子が存在するケースが数多く存在する。遺伝子重複と呼ばれるこのような系においては、一つの遺伝子に変異が起こって機能を失っても生存に支障が生じないため、変異した遺伝子の進化速度ははるかに速くなる事が知られているからである。実際、ゲノム解析の終わっている植物には非常に多くの類似したアミノ酸配列を担保する塩基配列が存在する。例えばシロイヌナズナでは、カルコンシンターゼとして検索すると75種の配列データが得られる。この結果を、アミノ酸残基数389から395 (392±3) の範囲に制限しても36種が残る。しかし、この36種の中で、タンパク質としての発現が確認されているものは1種に過ぎない。ブドウ(Vitis vinifera)に対して同じ検索をかけると、なんと264種の配列データが得られる。この結果にシロイヌナズナの場合と同様にアミノ酸残基数389から395 (392±3) の範囲に制限しても66種の配列情報が存在する。しかし、この中で転写レベルで確認されているものが15種、タンパク質としての発現が確認されているものはやはり1種に過ぎない。このような条件を考慮して、少なくとも転写レベルでは機能している事が確認されている遺伝子産物を使うという条件で進めることにした。
そこで、少なくとも転写レベルでの確認がなされているものを選んでみたところ910件のデータが得られた。前節で述べたゼニゴケのスチルベンシンターゼのアミノ酸残基数392との比較を念頭に置いて、アミノ酸残基数を389から395 (392±3) の範囲に制限してみた。すると31種の配列が得られるものの、スチルベンシンターゼの言葉で限定したため、スチルベンシンターゼに属するレスベラトロールシンターゼ、ピノシルビンシンターゼのグループが除かれてしまったため、これらについては改めて検索を行い、その結果を先のグループに付け加えることで35種の酵素のアミノ酸配列を得ることができた。とはいえ、この35種に問題がないわけではない。この配列を基に描いた樹形図を下に示したが、まず、この結果に含まれる植物の種類に偏りが大きすぎる。
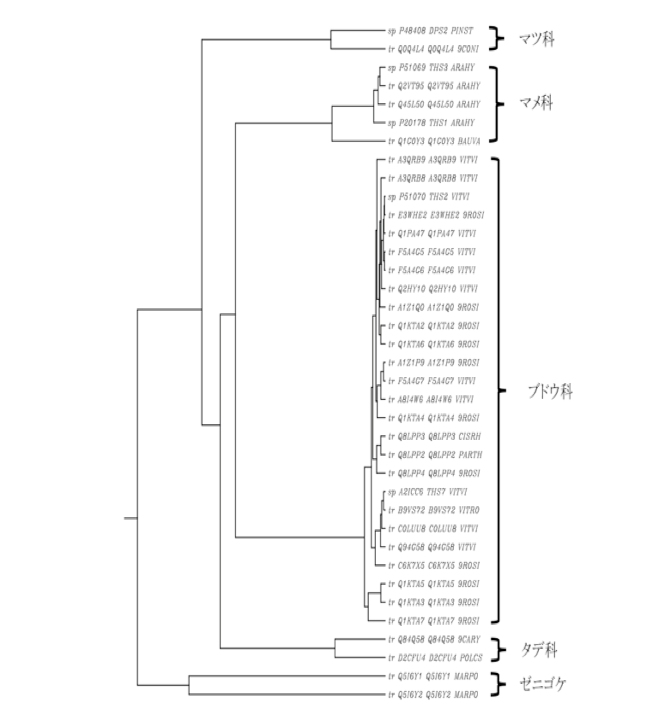
Rooted phylogenetic tree with branch length)
この集団の中には、先に述べたゼニゴケ(2種)の配列以外には、ブドウ科(26種)、マメ科(5種)、マツ科とタデ科(それぞれ2種)のスチルベンシンターゼのデータしか含まれていない。これで全植物をカバーした話ができるかと言われれば、首をかしげざるを得ないであろう。さらにブドウ科のデータ26種の酵素の中でいわゆるヨーロッパブドウ(Grape: Vitis vinifera)のスチルベンシンターゼのデータが12種を、中国の野生種ブドウであるVitis pseudoreticulataのデータが11種を占めている。
これはviniferaについては全ゲノムの解析が終わっていることが、pseudoreticulataについては、ウドンコ病耐性遺伝子の研究が活発に行われていることが原因であろう。しかしこれらのデータから高等植物界を網羅するような何らかの結論を導いたとしても,きっと誰も認めてはくれないだろう。私が読者の立場にあっても、顔を洗って出直してこいと言うに違いない。研究を続けているとこういう事はよく有ることで、こんな場合は筋が悪かったとあっさり諦めるのがよい。悩むに値しない事である。ではどうするか? 視点を変えればよいだけのことである。ここでとどまっても仕方ないので、カルコンシンターゼを含めもう少し大きな立場から考えを進めることにする。
ただし、この検索で、先に述べたゼニゴケ(Marchantia polymorpha)のスチルベンシンターゼ(脱炭酸を伴わずにルヌラリン酸合成において働くスチルベンカルボキシレートシンターゼ)とフタバネゼニゴケのカルコンシンターゼの配列が得られたのは幸運だった。
カルコンシンターゼについて
スチルベンシンターゼではどうもうまくいきそうにないので、気分を変えてカルコンシンターゼについて考えてみることにする。先に述べたように、カルコンシンターゼとスチルベンシンターゼはchalcone/stilbene synthases familyという同じ族に分類される酵素であり、アミノ酸配列だけでなく、基質との結合部位や活性部位に関するデータは、非常によく似てる。これは両酵素のアミノ酸の変異に対する制約がほぼ同じであることを意味しているため、カルコンシンターゼについて得られた進化速度は、スチルベンシンターゼについても適用できると推定した。この推定が間違っているとすれば以下の解析は無意味となる。
そこでスチルベンシンターゼの場合と同様に、アミノ酸残基数が389-395でかつタンパク質レベルで存在が確かめられているカルコンシンターゼを検索したところ、得られた結果はスチルベンシンターゼをキーワードにした場合と全く同じ結果になってしまった。どうやらUniProtというデータベースはカルコンシンターゼとスチルベンシンターゼをほとんど区別していないようである。
次に、カルコンシンターゼについて、そのアミノ酸残基数を389-395に限定し、かつ少なくとも転写レベルでの確認がされている条件で検索を実施したところ、351種の配列データが得られた。この351種のデータからchalcone synthase family protein あるいはchalcone synthase superfamily proteinなどと曖昧に記述してあるものを除くと、43科224種の植物に由来するカルコンシンターゼの配列データが残った。このグループは、43科224種とかなり広範な植物群を含むとともにデータ数もある程度確保できたため、データの中に少しのノイズが混入していても大筋では間違いのない結果が得られるのではないかと考え、このグループを用いて解析を続けることにした。
ではどのような比較をすればよいか。まず43科の内訳を見てみると、その殆どが被子植物であり、裸子植物ではイチョウ科とマツ科、シダ植物では、モクマオウのデータが存在する。これらの植物群の中で分岐時期が分かっているもの同士を比較すればいいのだが、証明したいカルコンシンターゼとスチルベンシンターゼの分岐時期に対して5億年近い値が予想されることを考えると、余りに近い時期に分岐したグループのデータから外挿することは好ましくない。そこで被子植物内部での比較はあきらめ、シダ植物、裸子植物、被子植物というグループ間での比較が適切であると考えた。
さて、陸上植物は維管束植物とコケ植物に大別される。維管束植物は、ヒカゲノカズラ類、シダ植物と種子植物からなり、陸上植物の中で最も原始的なグループであるコケ植物には、苔類、蘚類、ツノゴケ類からなっている。この3系統のコケ植物と、維管束植物の間の系統関係についてはよく分かっていなかったが、Qiu et al. (2006) は、陸上植物のなかで苔類が最初に分岐し、次いで蘚類そしてツノゴケ類が分岐したという結論を導いている。蘚類とツノゴケ類の分岐の順序についてはまだ異論がありそうであるが、苔類すなわちゼニゴケの仲間が最も古い陸上植物であることは、間違いなさそうである。そうであるなら、ゼニゴケが持つスチルベンシンターゼと維管束植物が持つスチルベンシンターゼあるいはカルコンシンターゼを比較することにより、この酵素群の進化速度と分岐時期が推測できることになる。
とはいえ、話はそう簡単には進まない。苔類とシダ植物、シダ植物と裸子植物はいつ分岐したのかについては、ある程度の情報が存在する。ところが被子植物については、裸子植物から分岐したのか、それともシダ植物から分岐したのかについてさえ確定していない。被子植物のデータが大部分を占めている解析において、被子植物の出現時期と起源が分からないではなにも始まらない。Darwinが、被子植物の起源の問題は Abominable mystery(忌まわしき謎)であると語っていたそうだが、私も「被子植物の起源」の問題を何とかしないと先には進めない。
古いというと語弊があるが、形態学的な立場から分類と起源を考えてきた研究者達の書籍を参照すると、藻類から被子植物へと進化する枝の途中で、ソテツ、イチョウ、針葉樹、グネツムの順に分岐していく系統樹が書かれている。しかも、それぞれがいつ分岐したかについては、古生代末期とか白亜紀の初期などとする曖昧模糊とした書き方しかされていない。これは非難をしているのではない。Aという植物とBという植物が分岐した瞬間の化石が都合良く残るはずはなく、ある程度の不確実さが残るのは当然であろう。もしあり得ないほどの僥倖に恵まれ、分岐した瞬間の化石が残ったとしても、これが分かれた瞬間の化石だと見分けること自体がそもそも不可能であるに違いない。しかしながら、DNA配列やアミノ酸配列の解析技術がいかに進歩しても、ある生物の出現の時期は化石記録との照合からしか得られない。そう言う意味において、正確な分岐の時期を求めるのは至難のわざであろう。とは言え、分岐した時期を正確につかまえない限り、分子時計は正確な値を示さないであろう。
さらに、まだ問題は存在する。カルコンシンターゼ、スチルベンシンターゼともに、野生植物からのデータはきわめて少ない。いわゆる研究者と呼ばれるある意味でオタクな人種であっても、花や葉に特徴がなく、果実が美味しいわけでもない、毒にも薬にもならない野生の植物から、こうした酵素のデータを取るような物好きはまず存在しない。産官学の共同研究が叫ばれ、科研費においてもいつどのように役立つかを嘘でも良いから歌い上げないと採用されない現在では、役に立つかどうか分からないそんな研究に予算を出してくれる組織はまず存在しない。(言葉の使い方だが、科研費すなわち科学研究費は、その名称を技術研究費と改名した方が実態に合っている。偽研費ならもっと正確だ)従って、多くのデータは栽培植物由来のものとならざるをえない。そうすると、人為的な選択が大きな問題となる。特に花、葉、果実を重視する栽培植物においては、ヒトによる進化への介入があったに違いない。結論としては、ある程度の誤差を受け入れると同時に、新しい知見をできるだけ取り入れた形で進めてみるしかないということになる。
従来、藻類から被子植物に向かう植物の進化は、下図のAに示すようにまずソテツが分岐し続いてイチョウ、針葉樹、グネツムの順に分岐したと考えられてきた。
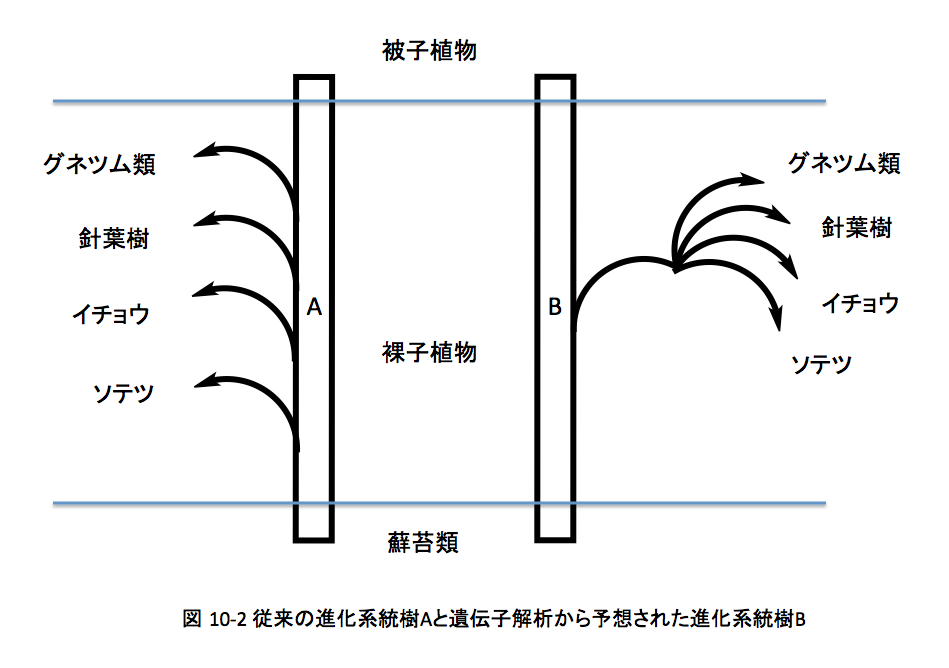
しかし近年では、DNAの解析結果からこの系統樹の信憑性に疑問符がついた状態にある。そこで本解析では、長谷部氏らがMADSボックス遺伝子(後生動物、菌類、緑色植物に分布する転写因子)の解析から描いた図10-2に示したBの系統樹を採用した。この系統樹においては、被子植物と裸子植物であるソテツ、イチョウ、針葉樹、グネツムの進化距離が同じであるという点で、従来の形態学的指標から推定された系統樹と大きく異なっている。次に、この系統樹における各植物の分岐時期を推定しなければならない。シダ植物から被子植物と裸子植物の共通祖先であるシダ種子植物が分岐した時期については、古生代デボン紀の終わり頃とされている場合が多い。従って、シダ種子植物については、その出現時期を3.6億年前という値を採用した。裸子植物と被子植物の分岐時期については、従来は白亜紀とジュラ紀の境界付近にある(1.4億年前)とされてきたが、三畳紀にまで遡る可能性も語られてきた。近年、遺伝子解析による被子植物の系統分類を行うグループ(APG)が1.8億年という値を提案している。この値はジュラ紀の前期までその出現を繰り上げる提案であり、私もこの値を採用することにした。
あれこれといろんな条件が重なって、話の道筋が分かりにくくなっているが、これは読者に責任があるのでなく、一部は書いている筆者の混乱が現れているにすぎない。ただ、物事を整理しようとするとき、こうしたカオス的状況になることは良くあることで、大掃除の途中であると考えればよい。そこで、ぐだぐだと条件を述べるのではなく、実行すべき目的に対してどう考え何をすべきかを箇条書きにしてみた。
1. カルコンシンターゼとスチルベンシンターゼの進化速度は同一として扱う。
2. カルコンシンターゼについての解析を行う。
3. 解析には、43科220種の植物に由来し、少なくとも転写レベルでの確認がされているカルコンシンターゼのアミノ酸配列データを使用する。
4. シダ植物(モクマオウ)、裸子植物(マツ属4種、モミ属2種、イチョウ属2種、)と被子植物(43科227種)について、それぞれ置換されたアミノ酸残基数を求め、アミノ酸1残基あたりの進化速度を算出する。
5. シダ植物から被子植物と裸子植物の共通祖先であるシダ種子植物が分岐した時期については3.6億年を採用する。
6. 裸子植物と被子植物の分岐時期については1.8億年を採用し、針葉樹とイチョウの分岐時期は同じとする。
7. ゼニゴケのスチルベンシンターゼと高等植物のカルコンシンターゼを比較して変異を起こしたアミノ酸残基数を求め、この変異アミノ酸残基数から両酵素 (両植物) の分岐時期を推定する。
8. 全体の解析を通して、酵素遺伝子の進化と植物の進化に対応性があるものと仮定する。(遺伝子の水平移動などは起こっていないと考える)
以上の条件と仮定の下に、解析を行うことにする。
