そこで、実際の解析に入るわけだが、この段階で頭を悩ませる事実がいくつか出現した。まず、ある程度信用できる、タンパク質としての発現が確認されているカルコンシンターゼについて述べることにする。
カルコンシンターゼについて
UniPlot上でタンパク質として発現が確認されているカルコンシンターゼは以下の5種類である。一寸だけアミノ酸の並びを紹介する。もちろん無視されても結構です。
>sp|P30074|CHS2_MEDSA Chalcone synthase 2 OS=Medicago sativa GN=CHS2 PE=1 SV=1
MVSVSEIRKAQRAEGPATILAIGTANPANCVEQSTYPDFYFKITNSEHKTELKEKFQRMC
DKSMIKRRYMYLTEEILKENPNVCEYMAPSLDARQDMVVVEVPRLGKEAAVKAIKEWGQP
KSKITHLIVCTTSGVDMPGADYQLTKLLGLRPYVKRYMMYQQGCFAGGTVLRLAKDLAEN
NKGARVLVVCSEVTAVTFRGPSDTHLDSLVGQALFGDGAAALIVGSDPVPEIEKPIFEMV
WTAQTIAPDSEGAIDGHLREAGLTFHLLKDVPGIVSKNITKALVEAFEPLGISDYNSIFW
IAHPGGPAILDQVEQKLALKPEKMNATREVLSEYGNMSSACVLFILDEMRKKSTQNGLKT
TGEGLEWGVLFGFGPGLTIETVVLRSVAI (アルファルファ:マメ科)
>sp|P13114|CHSY_ARATH Chalcone synthase OS=Arabidopsis thaliana GN=CHS PE=1 SV=1
MVMAGASSLDEIRQAQRADGPAGILAIGTANPENHVLQAEYPDYYFRITNSEHMTDLKEK
FKRMCDKSTIRKRHMHLTEEFLKENPHMCAYMAPSLDTRQDIVVVEVPKLGKEAAVKAIK
EWGQPKSKITHVVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRIAK
DLAENNRGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFSDGAAALIVGSDPDTSVGEK
PIFEMVSAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIVKSLDEAFKPLGISD
WNSLFWIAHPGGPAILDQVEIKLGLKEEKMRATRHVLSEYGNMSSACVLFILDEMRRKSA
KDGVATTGEGLEWGVLFGFGPGLTVETVVLHSVPL (シロイヌナズナ:アブラナ科)
>sp|P23569|CHSY_PUEML Chalcone synthase OS=Pueraria montana var. lobata GN=CHS PE=1 SV=1
MVSVAEIRQAQRAEGPATILAIGTANPPNCVDQSTYPDYYFRITNSEHMTELKEKFQRMC
DKSMIKKRYMYLTEEILKENPNMCAYMAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQP
KSKITHLIFCTTSGVDMPGADYQLTKQLGLRPYVKRYMMYQQGCFAGGTVLRLAKDLAEN
NKGARVLVVCSEITAVTFRGPSDTHLDSLVGQALFGDGAAAVIVGSDPIPQVEKPLYELV
WTAQTIAPDSEGAIDGHLREVGLTFHLLKDVPGIVSKNIDKALFEAFNPLNISDYNSIFW
IAHPGGPAILDQVEQKLGLKPEKMKATRDVLSDYGNMSSACVLFILDEMRRKSAENGLKT
TGEGLEWGVLFGFGPGLTIETVVLRSVAI (クズ:マメ科)
>sp|Q9FUB7|CHSY_HYPAN Chalcone synthase OS= PE=1 SV=1
MVTVEEVRKAQRAEGPATVMAIGTAVPPNCVDQATYPDYYFRITNSEHKAELKEKFQRMC
DKSQIKKRYMYLNEEVLKENPNMCAYMAPSLDARQDIVVVEVPKLGKEAAVKAIKEWGQP
KSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAEN
NKGARVLVVCSEITAVTFRGPTDTHLDSLVGQALFGDGAAAIIIGSDPIPEVEKPLFELV
SAAQTILPDSEGAIDGHLREVGLTFHLLKDVPGLISKNVEKSLTEAFKPLGISDWNSLFW
IAHPGGPAILDQVEAKLSLKPEKLRATRHVLSEYGNMSSACVLFILDEMRRKSKEDGLKT
TGEGIEWGVLFGFGPGLTVETVVLHSVAIN (コボウズオトギリ:オトギリソウ科)
>sp|Q8RVK9|CHS_CANSA Naringenin-chalcone synthase OS=Cannabis sativa GN=CHS PE=1 SV=1
MVTVEEFRKAQRAEGPATIMAIGTATPANCVLQSEYPDYYFRITNSEHKTELKEKFKRMC
DKSMIRKRYMHLTEEILKENPNLCAYEAPSLDARQDMVVVEVPKLGKEAATKAIKEWGQP
KSKITHLVFCTTSGVDMPGADYQLTKLLGLRPSVKRLMMYQQGCFAGGTVLRLAKDLAEN
NKGARVLVVCSEITAVTFRGPNDTHLDSLVGQALFGDGSAALIVGSDPIPEVEKPIFELV
SAAQTILPDSDGAIDGHLREVGLTFHLLKDVPGLISKNIEKSLNEAFKPLGISDWNSLFW
IAHPGGPAILDQVESKLALKTEKLRATRHVLSEYGNMSSACVLFILDEMRRKCVEDGLNT
TGEGLEWGVLFGFGPGLTVETVVLHSVAI (アサ:アサ科)
単純だがこの配列を基に系統樹を描くと次の系統樹(図10-3)が得られる。この結果をゲノム解析を基本に植物を分類するAPG II(APG植物分類体系 2003年に発表された第2版)の結果と合わせると、アサの入る位置がずれてはいるが、残りは一致する。

まあ左程悪くはない結果だと次に進もうとしたとき、一寸まずいデータを見つけてしまった。ゲノム解析が終わっているいくつかの植物で予備的に見てみようと思ったのが原因である。まず、シロイヌナズナのカルコンシンターゼをみてみたら(sequence length 384~400)、なんと38個の配列が引っかかってきた。関係がないわけではないが、一寸関係なさそうな1個を除き、残りの37個のタンパク質について系統樹を描くと図10-4のようになる。
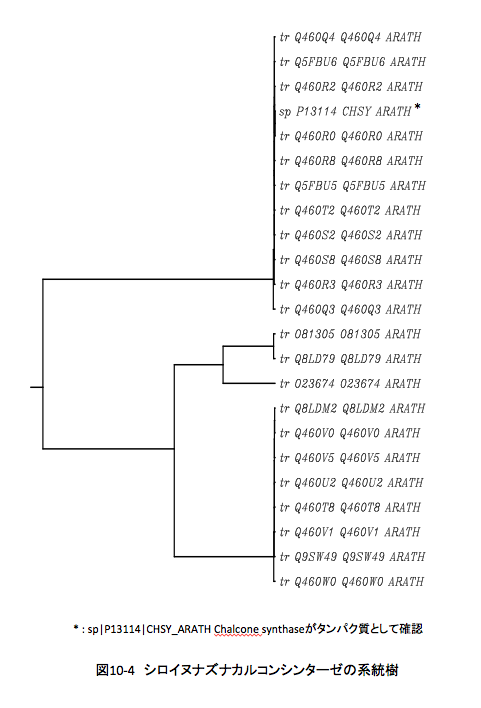
見て分かるように、大きくみれば2つのクラスターに分かれるのだが、タンパク質として発現しているカルコンシンターゼは上から4段目にあるsp P13114 CHSY ARATHだけである。このタンパク質を含むクラスターともう一つのクラスターの変異は異常に大きい。これをどう評価するか。コップの中の混乱をコップの中だけで見ていては埒はあかない。適当にと言うと余り適切でない言葉の選択になるが、適切にと言うと選んだ基準の明確化が求められる。その中間辺りの判断で、その他の植物(被子植物8種、裸子植物2種、シダ植物1種)を選んで、これらの植物で転写が認められている配列を加えて系統樹を描いてみた。その結果を図10-5に示す。
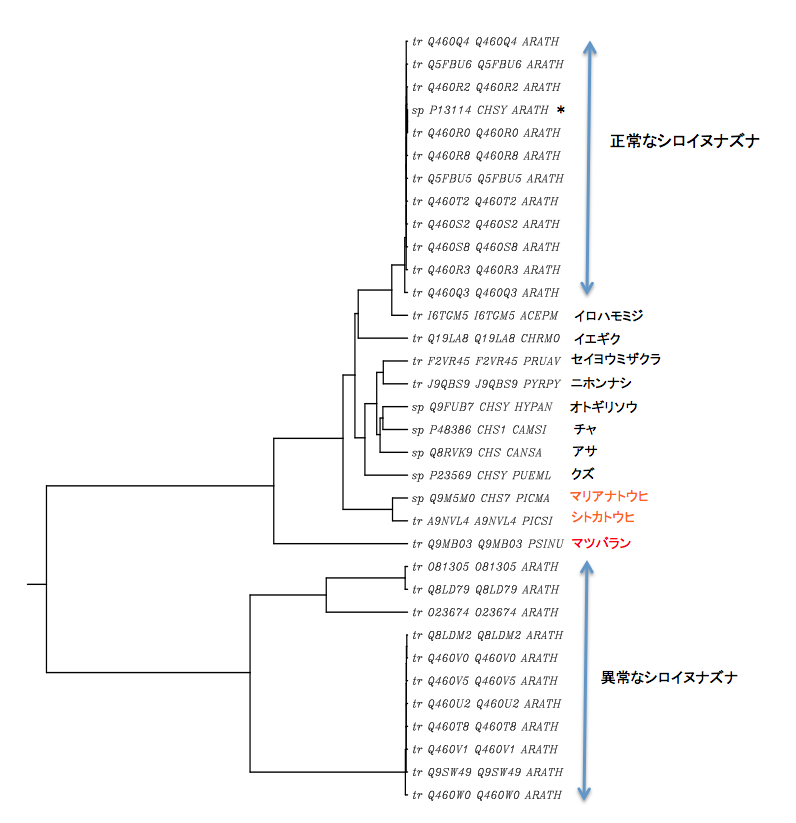
ターゼを比較した樹形図
見て明らかなように、シロイヌナズナの異常なタンパク群は完全に独立したクラスターとなるわけだが、正常なタンパク群との変異の程度は、被子植物や裸子植物どころかシダ植物のマツバランをはるかに越えるものである。この図が分岐してからの時間を枝の長さに反映させて描くUPGMA法に依っていることを考慮すると、正常群と異常群のタンパク質が分かれたのはシダ植物とその他の植物が分岐した3.6億年の2倍以上昔の話となり、カルコンシンターゼが高等植物にしか分布しないという結果と矛盾することになる。さらに、適切に(?)選んだその他のタンパク質の全てが、正常なシロイヌナズナと同じクラスターに属することを考えれば、この異常群のデータは除外すべきであると判断せざるを得ない。よく似た現象が、Arabis gemmifera(ハクサンハタザオ)、ヨーロッパブドウ(Vitis vinifera)、ホップ(Humulus lupulus)あるいはペチュニア(Petunia x hybrida)などで見られる。これらの異常なタンパク質群は、苔類までの植物が持っていたスチルベンシンターゼがカルコンシンターゼに進化した後、遺伝子の重複に伴ってその中のいくつかが偽遺伝子と化し、酵素機能という拘束からはずれて高い速度で進化した現象を反映したものであろう。この現象については、後でもう少し詳しく述べる予定である。
そこで改めてUniPlotにおいてchalcone synthase で検索した後、タンパク質レベルあるいは転写レベルで確認されていること、一寸範囲が大きくなったがアミノ酸残基数386-400、かつフラグメントではないことを条件に絞り込みを行ったあと、APG IIの結果等を加味しながら明らかに異常と思われるシークエンス除去した結果、44科に渡る植物のカルコンシンターゼ95種を選抜することができた。これに、タンパク質レベルでの発現が確認されている5種を加えた100種の配列を解析に使用することにした。
この時、イチョウから2種、マツ科植物から14種、マツバラン(シダ植物)1種のカルコンシンターゼのアミノ酸シークエンスも得られた。従って、これらの植物間の配列を比較することで、まずカルコンシンターゼの進化速度を求めることにした。
「国境のトンネルを抜けると北国であった」とは川端康成の雪国の冒頭であるが、カオスの海から抜け出すことができれば、新しい地平が見えるかもしれない。
アミノ酸配列の解析にはNCBI/BLASTを用いたが、計算に使用するカルコンシンターゼの選抜に長い時間をかけたというのに、計算にかかる時間はほんの十数秒、単純作業に対する計算機の仕事の速さは異常に早い。AIに負けるかもしれないなと思いつつも、発想の伸びというか奇抜さではまだ負けないと思っている。そこでだが、シダ植物であるマツバランのカルコンシンターゼ(CHS)と被子植物100種のCHSについて比較した結果を例にして、計算方法等の説明をする。この解析では、マツバランのCHSのアミノ酸配列と被子植物1種のCHSのアミノ酸配列を比較し、比較に使用したアミノ酸配列部分のアミノ酸座位数と変異の起こったアミノ酸座位数を求める。この解析を残りの99種のCHSに対して行うと、総計100個の解析データが得られる。これらをバラバラに計算してもしかたがないため、 解析に用いたアミノ酸座位数と変化の起こったアミノ酸座位数の全てについて、それぞれ平均を取ると、前者の対しては384.5、後者に対しては122.3という値が得られた。(この段階で平均を取って良いかどうかについては、数学的意義付けにおいて、一寸だけ不安がないわけではない)ともあれ、これは384.5個(標準偏差σ=0.9)のアミノ酸残基において、シダ植物と被子植物(高等植物)との間に122.3個(標準偏差σ=2.9)の変異した残基が存在することを示すとともに、少なくとも2つの母集団がかなりまとまりの良いデータ群であることを意味している。
さて、この二つの植物が分岐したのが3.6億年前であるから、このデータを基に、安易に1アミノ酸残基・1年あたりの進化速度を
(122.3/384.5)÷(3.6×108)÷2 = 0.44×10-9
としてはいけない。(最後に2で割ったのは、共通の祖先から片方はモクマオウへの進化、もう一つは高等植物への進化があり、二つの変化の和がアミノ酸残基122.3座位の変化として観察されているためである。) 比較する酵素のアミノ酸配列が、系統的に近縁である場合(変異数が少ない場合)は同一の座位での重複置換を考慮する必要はないが、384.5部位あたり122.3もの部位で変化が起こっているとすれば、同じ部位で重複の変異が起こっている可能性に基づく補正が必要となる。補正の計算方法については、培風館から出版されている「分子進化学入門」を初めとする分子進化の中立説に関する書籍等を参照して欲しいが、現在では安田徳一という方がウェブ上に「集団遺伝学講座」という丁寧な記事を上げておられるので、ここを参照されるのがいいだろう。https://www.primate.or.jp/old/PF/yasuda/38.html
私の数学的能力は、それほど高くない。高校程度の数学まではOKだったが、大学の数学となるとちょっと抽象的すぎて、頭を抱えた経験が一度ならずあった。さて、タンパク質分子の中で基質の認識部位や活性部位などほとんど変化できない部位を除けば、アミノ酸の部位あたりの置換数の分布はポアソン分布に従うと仮定する。ここまでは何とか理解できる。ポアソン分布、正規分布に似ているがサンプル数が少ないときに用いられるもので、はかない記憶を辿ればフランスの軍隊で、ウマに蹴られて死ぬ兵士数の分布だったやつだ。計算法については何の記憶もないくせに、こうした余談だけは覚えている。学生とはそう言う奴らであることをよく認識して講義をすべきだとつくづく思う今日この頃であるがもう遅い。要するに、分布の基礎になったサンプルが意外だったが故に何とか記憶していただけのものである。
さて、分化前のタンパク質が二つのタンパク質に向かって分化する過程で、一つのアミノ酸部位で平均Kaa回の置換が起こったとする。アミノ酸の総部位数をnaaとすると、一つの部位に置換が1、2、3・・・回起こる予測数は
naaexp(-Kaa)、naaKaa exp (-Kaa)、naa(Kaa2/2)exp(-Kaa)、・・・
で表されるから(というところでちょっと分からなくなり)、それぞれに実測した置換の数から未知のパラメータ(-Kaa)を推定することができる。(そうですか)(-Kaa)ここでさらに変異が起こる確率は全ての部位で同じであると仮定する。この仮定を外すとポアソン法則が成立しなくなり(例えば負の2項分布法則)・・・と書いてあるところで完全にお手上げになる。話の筋道は何とか理解できるが、式を立てて計算ができるかと問われれば、御免なさいというほかはない。まあこの部分は数学の得意な先達にお任せして,最終の式を使わせて頂くことにする。
まず、1アミノ酸部位あたりに起こった置換の回数をKaaとする。ここで、全アミノ酸の部位数をnaa、アミノ酸の違いが観察された部位数をdaaとすると、naa- daaは変異が観察されなかった部位の数となる。変異のなかったアミノ酸部位について、観察数と予測数が等しいと置くと
naa – daa = naaexp(-Kaa)
であるから、Pd = daa /naaとおいて、両辺の対数をとれば次の式からKaaを求めることができる。
Kaa = -ln(1-Pd) = -2.3log(1-Pd) ・・・・・(この程度なら問題なく分かる)
なお、ここで求めたKaaは統計学でいうところの最尤推定値である。ここでKaaに対する標準誤差σKは次の式で求められる。
σK = √[Pd/{(1–Pd)naa}]
この式にnaa=384.5 daa=122.3を代入すると、Kaa= 0.384±0.035が得られる。こうして推定したKaaは、2つのタンパク質が共通の祖先から分化する過程で、それぞれT時間の間に置換したアミノ酸の部位当たりの総数を意味し、合計2T時間にわたる変化である。従って、単位時間(年)あたり、1アミノ酸部位当たりの置換率(言い換えれば進化速度)はkaaは
kaa=Kaa/(2T)
で求められる。ここで、シダ植物と被子植物が分岐したのが3.6億年前と推定されているので
kaa=(0.53±0.049)×10-9
という値が得られる。 続いて、マツバランと裸子植物(13種)との比較から求めたカルコンシンターゼ進化速度を示している。この場合も両植物の分岐時期は3.6億年前である。従って得られるkaa 値は(0.51±0.045)×10-9となり、この値は先に求めたシダ植物-被子植物間で求めた値と、同一と考えて良い。この結果に気をよくして、13種の裸子植物に由来するカルコンシンターゼをそれぞれ100種の被子植物由来のカルコンシンターゼと比較してみた。この場合は2種類の植物の分岐が1.8億年前であることより、Tの値に1.8x108の値を代入している。そうすると表に示しているように、全ての場合においてkaaは(0.50 ± 0.023)×10-9から(0.54 ± 0.023)×10-9の間に収まっている。これらの値はまた、先ほどのシダ植物と裸子植物/被子植物間で得られた値と同じである。下に示す表12-1にこの結果を示す。
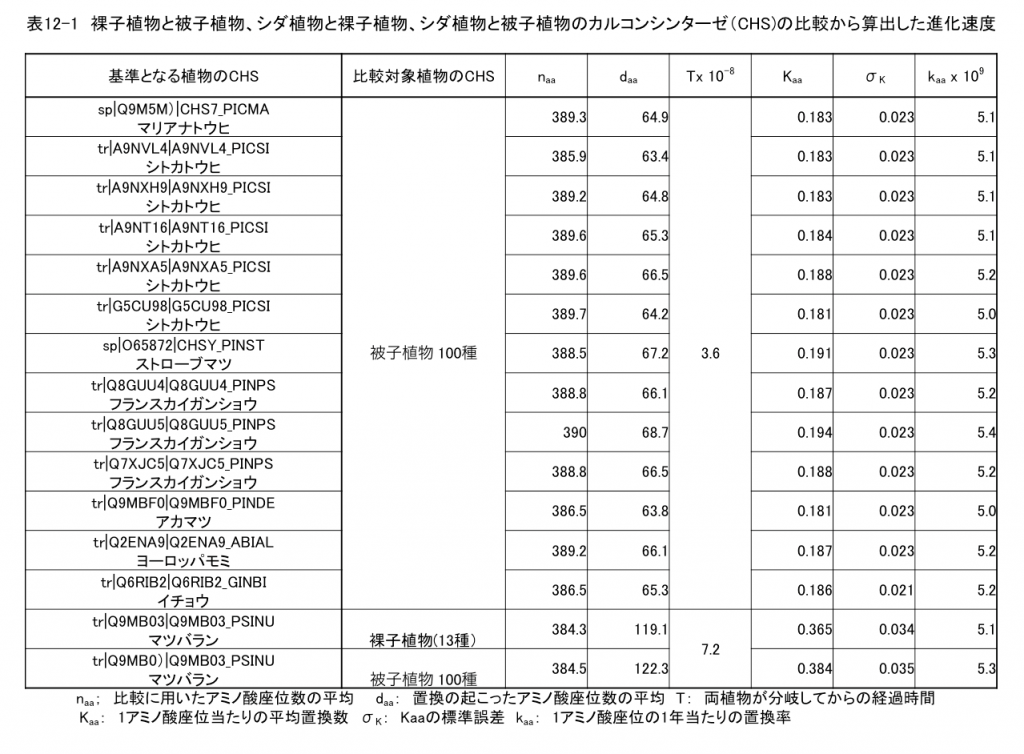
実際に解析を始める前には、ここまできれいに値が揃ってくるとは期待していなかった。思いがけないほどの結果である。
では、これらの値がタンパク質としての進化速度として妥当であるかどうかを考えてみよう。タンパク質の進化速度は、そのタンパク質の構造に対する制約の強さに依存している。タンパク質の中で、アミノ酸配列に対する制約が殆どないフィブリノペプチドは進化速度が速いことで知られているが、その kaa 値は8.3×10-9である。一方、進化速度の遅いタンパク質としてはチトクロームCやヒストンH4などが存在し、それらのはkaa 値はそれぞれ0.3×10-9、0.01×10-9であることが知られている。上記の値はこの範囲に収まっており、異常な値ではない。一般的な進化速度を持つタンパク質であるインスリンやミオグロビンの進化速度と比べると、インスリン(kaa=0.44×10-9)よりもいくぶん早く、ミオグロビン(kaa=0.89×10-9)よりも遅い程度であり、酵素の進化速度として妥当な値であると考えられる。
どうやら、カルコンシンターゼという酵素は、1年1アミノ酸座位あたりおおよそ0.52x10-9という早さで置換が起こるようである。言い方を変えれば、カルコンシンターゼは概ね400万年に1残基程度の割合で変化してきた事を意味している。また、スチルベンシンターゼとカルコンシンターゼは同じグループに属するする酵素であるため、スチルベンシンターゼにおいてもカルコンシンターゼと同じ早さで進化すると考えた。
こんな書き方をしてはいるが、初めに計算を志したときに、ここまできれいなデータが得られるとは思っていなかった。被子植物内での比較から進化速度を出そうともがいていた頃は、値のばらつきに頭を抱えたものである。団塊の世代の一人として、若い頃にヘーゲルの著作を繙いたことがある。特に共感して読んだわけではなく、若者特有のスノビズムのなせることであったが、こうしたデータを眺めていると、『現象は体系である』とする彼の思想が思わずよみがえってくる。
一寸待て、このブログはアブシジン酸について書くとしていながら、全くアブシジン酸が出てこないではないか。「アブシジン酸で検索して辿り着いたのに、アブシジン酸は出てこない。羊頭狗肉だ」などとする感想をお持ちの方がいないとも限らない。確かに、スチルベノイドとフラボノイドの話で本人は盛り上がっている。そこにはアブシジン酸の姿はないように見える。しかし、スチルベノイドの話は、隠れたルヌラリン酸の話である。スチルベノイドの生合成を行うスチルベンシンターゼは、高等植物には原則として存在しない。つまり、ルヌラリン酸は高等植物には原則的には存在しない。では、下等植物においてルヌラリン酸が果たしていた役割、つまりルヌラリン酸のニッチを受け継いだのはアブシジン酸ではないかという議論をしているわけである。
「ひとつの天然物の総合的理解においては、その化合物に関わる周辺化合物の理解が必須である。」と筆者は考える。近年のような業績原理主義の時代においては、周辺の知識を得るための努力は殆ど評価されない。簡便なキットを使って、小さな事柄でも良いから新規なデータを出した方が、ノートでも良いから報告を書いた方が、就職においても昇進においても遙かに有利である。だが、それだけで良いのだろうか?ルヌラリン酸あるいはアブシジン酸のみを詳細に研究したとしても、それらの真の姿は分からない。ある化合物は、周辺の化合物群との相互比較によって、その存在意義が顕在化する。無根系統樹に根をつけるには、アウトグループすなわち解析しているグループとは違うグループに属する生物のデータが必要ではないか。
とはいうものの、少しばかり論点を整理しよう。筆者は、植物が進化に際してその生育抑制型生長調節物質をルヌラリン酸からアブシジン酸に変更したと考えた。この変更を支持する化学的・生理学的データについては、両化合物の化学的、生理学的類似性については「ルヌラリン酸とアブシジン酸」で論じた。要するに、両化合物は非常によく似た立体構造を持ちうるだけでなく、同一の遺伝子の発現を通して同じ生理現象を引き起こす。ここまでの話には矛盾はない。
しかし、ルヌラリン酸はゼニゴケ類以下の下等植物に分布し、アブシジン酸はスギゴケ以上の高等植物に存在するという事実について、現在のところ合理的な説明はされていない。アブシジン酸はアブシジン酸であり、ルヌラリン酸はごくマイナーな下等植物の生長調節物質であるに過ぎない。ここに歴史的な視点を取り込んだアブシジン酸によるルヌラリン酸のレセプター乗っ取り仮説を押し込んだわけである。もっとも、その仮説自体が私の個人的な意見に過ぎず、学会で認知される状況になっていないのだから、私が合理的な説明をしない限り誰もするはずはない。自明のことである。
図10-6にアブシジン酸とルヌラリン酸だけでなくフラボノイドとカロテノイドに属する化合物群について、関連する事項を整理してみた。これを基に話を続けよう。アブシジン酸については、藻類(オステロコッカス・クラミドモナス・ボルボックス)にはビオラキサンチンまでの生合成酵素しか存在しない。C20-C21の間の結合を切断する酵素は蘚類(ヒメツリガネゴケ)以上の高等植物において機能している様である。(KEEG参照)ゼニゴケ類についてのデータはまだ得られていないにしても、アブシジン酸はスギゴケ以上の維管束植物に分布するという言明は、実験的結果は別にして、この事実に依拠していると考えて良い。
一方、ルヌラリン酸は苔類以下の下等植物に分布する。ルヌラリン酸が含まれるスチルベノイドと呼ばれる化合物群は、スチルベンシンターゼによって生合成されるのだが、このスチルベンシンターゼは一部の例外を除くと高等植物には分布しない。ルヌラリン酸については、それで矛盾はない。しかしながら、ルヌラリン酸が植物ホルモン的に機能しているとすれば、この段階でのスチルベンシンターゼの進化によって、このホルモンのニッチが空席になることを意味する。ここで2つの問題が生起する。1つは、“このニッチを継承する化合物は何か”という問題であり、いま1つは、“スチルベンに向かって流れていた代謝はどこに向かったのか”という問題である。
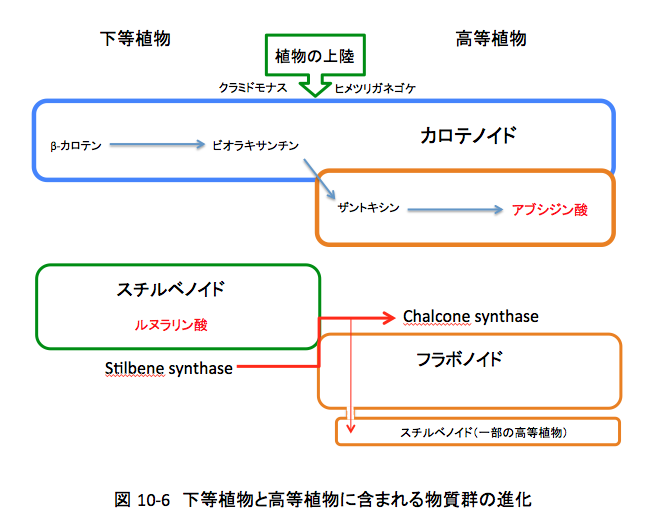
第1の問題には、ルヌラリン酸のニッチをアブシジン酸が継承したとする暫定的な答えを提出している。何故そう考えるのかという理由については後でもう少しだけ議論することにする。第2の問題が、この章で取り上げている問題である。図10-6には示していないが、クラミドモナスとヒメツリガネゴケの間に苔類のゼニゴケが存在する。その苔類にはスチルベンだけではなく、フラボノイド類であるフラバノン、フラボン、フラボノールが存在する。ゼニゴケの仲間であるフタバネゼニゴケにおいて、CHS-like proteinとされている2本の配列が知られているが、これらが本当にCHS活性を持つのかSTS活性を持つのかは不明である。さらに、蘚類に属するヒメツリガネゴケ((Physcomitrella patenssubsp. patens)subsp. patens)はゲノム解読が終わっている植物である。この植物について、UniPlotを用いてChalcone synthaseとPhyscomitrella patensをキーワードにして検索すると27種のアミノ酸配列が得られる。ところがキーワードをStilbene synthaseとPhyscomitrella patensの組み合わせにしても、同じ29種の配列が得られる。この中にはアミノ酸残基数が500前後の3-ketoacyl-CoA synthaseとされる配列12種が含まれる。この酵素群は脂肪酸やポリケチドの生合成で働く酵素で、反応メカニズムはCHS・STSと同じと考えて良い。さらに、CHS・STSとアミノ酸配列においてもかなりな相同性があることを考えれば、多分この3-ketoacyl-CoA synthaseがCHS・SSの祖先であろう。
しかしいま、3-ketoacyl-CoA synthaseまで視野に入れると、視野が広がりすぎて話が混乱しそうである。そこで、これらの配列は横に置くだけでなく、幾つかのフラグメント配列を除外すると10種のアミノ酸配列が残ってくる。現時点では、これらのCHSと予想される配列の中に、タンパクレベルでの発現が認められているものは無いし、転写レベルでの発現しているものも知られていない。だが、同じPhyscomitrella patensの他株から転写レベルで働いているCHSのアミノ酸配列が得られている。この配列 tr Q2VAZ3と11種の配列を比較すると、図10-7に示すようにtr A9U1W8とtr A9RQT8のアミノ酸配列がほとんど同じである。従って、この2つのタンパク質のいずれか、あるいは両方がこのコケの中で機能していると仮定しても良いだろう。

少し更新が遅れた理由は、ゼニゴケのSTSはスチルベンシンターゼと確定しているものの、フタバネゼニゴケのCHS、ヒメツリガネゴケのCHSと推測されている配列群について、それらが本当にCHSであるのか、それともSTSであるのかを、確認できないかと足掻いていたことも原因である。しかしこれは無理なように思える。余り微細なことにこだわるより、ここは少しあらっぽく、下等植物はスチルベン類を含み高等植物はフラボノイドを含むと把握した方が、良さそうである。そうすると、ゼニゴケの仲間である苔類が、いまの高等植物の直接の先祖ではないにしても、その分岐点に位置し上陸を敢行した植物群であり、この上陸と時期を同じくして、STSからCHSへの進化が起こったと考えたら、どのような結果が得られるか。
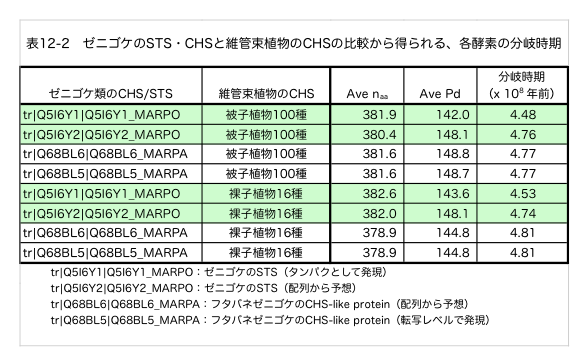
ゼニゴケにおいては、タンパク質レベルで発現している392残基からなるSTS(tr|Q5I6Y1|Q5I6Y1_MARPO Stilbenecarboxylate synthase 2 OS= Marchantia polymorpha)とアミノ酸配列からSTSと予想される393残基からなるタンパク質(tr|Q5I6Y2|Q5I6Y2_MARPO Stilbenecarboxylate synthase 1 OS=Marchantia polymorpha)がある。この2本のアミノ酸配列を、先の解析で使用した100種の被子植物のCHS、16種の裸子植物16種のCHSと比較すると、Q5I6Y1_MARPOと維管束植物についてはnaa=381.9、daa=142.0、裸子植物に対してはnaa=382.6、daa=143.6という値がえられる。同じくQ5I6Y2_MARPOに対しては、それぞれnaa=380.4、daa=148.1、naa=382.0、daa=148.1という値が得られる。それぞれの値から分岐時期を推定すると、それぞれ4.48億年前と4.53億年前、4.76億年前と4.74億年前という値が得られる。これらの値に有意差があるかと問われれば首をかしげざるを得ない。kaaの誤差を考えれば、ほぼ同じと考えて良いのかもしれない.簡単に言えば、維管束植物のカルコンシンターゼとゼニゴケのスチルベンシンターゼは4.5~4.8億年前に分岐したといえる。
しかし、たったこれだけの例で間違いないと断言する訳にはいかないだろう。そこで適切な例はもうないかと考えた。先にゼニゴケの仲間であるフタバネゼニゴケにはCHS-like protein として分類される2本のアミノ酸配列があることを述べた。実際のところ、これらがCHSであるのかSTSであるのかは分からないが、1つは転写レベルでの発現が認められているタンパク質(tr|Q68BL6|Q68BL6_MARPA Chalcone synthase-like OS=Marchantia paleacea subsp. diptera)、いま1つはアミノ酸配列からCHS-like protein と予想されているタンパク質(tr|Q68BL5|Q68BL5_MARPA Chalcone synthase-like OS=Marchantia paleacea subsp. diptera)である。そこで、この2種のタンパクを先の解析で使用した100種の被子植物のCHS、16種の裸子植物16種のCHSと比較すると、これらのタンパク質は4.77億年前に被子植物のCHSと、4.81億年前に裸子植物のCHSと分岐したとする結果が得られる。そしてこれらの値は、先の結果とほぼ同一であると考えて良いだろう。。図と表のナンバリングがおかしいのは、以前のブログで使っていた章立てが残っていたせいである。変更が面倒なのでそのままにして使っているので、気にせずに見て欲しい。
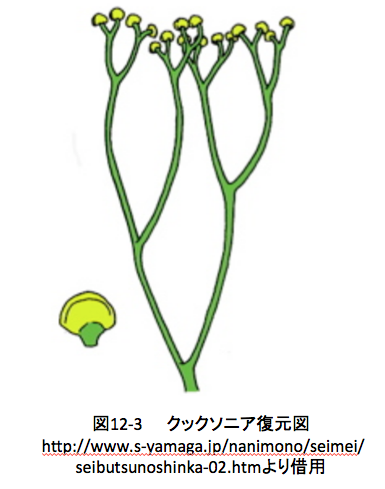
ではそこから、どのようなストーリーが描けるのか。ここで、形状が確認できる最も古い陸上植物の化石は、4億1000万年前(シルル紀中期)のクックソニアの化石である。クックソニアはコケ類から進化した植物で、維管束は持たないものの地上からは立ち上がっている.苔類に近い植物の実際の上陸はこれよりも早く、オルドビス紀(約5億0900万年前から約4億4600万年前)まで遡るのではないかと考えられている。さらに、コケ類の胞子ではないかと見られている微化石が、4.7億年前の地層から発見されている。(Wellman CH, Gray J., The microfossil record of early land plants. Philos Trans R Soc Lond B Biol Sci. 717–732 (2000)
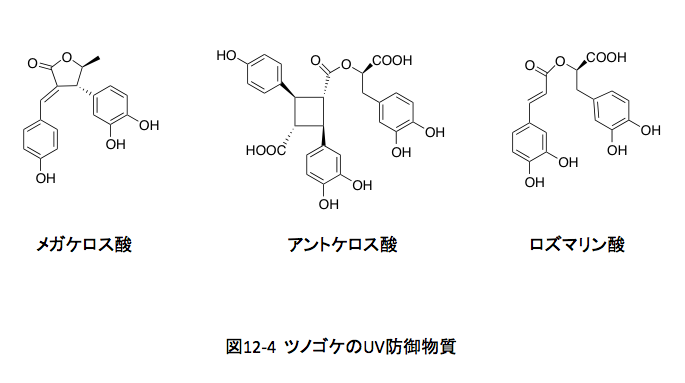
そうすると、先に得られた値は、丁度そのオルドビス紀の中期に相当する。オゾン層はすでに成立していたとはいえ、強い紫外線の照射下で上陸を始めた植物たちが、地上での生き延び方を模索していた時代であったろう。苔類においては300 nm付近にブロードで強い吸収を持つスチルベン類を用いて適応を図ったように思われる。ツノゴケ類は、その起源に関して分かりにくい部分をもつ植物だが、彼らはスチルベンやフラボノイドを使わずに、図12-4に示すようなメガケロス酸、アントケロス酸、ロズマリン酸などのリグナンをUV防御物質に使って進化してきたようだ。そして、現代の維管束植物に連なる原始陸生植物は、まさにこの時点で彼らが持っていたスチルベンシンターゼをカルコンシンターゼへの進化が起こったがゆえに、紫外線防御を担う物質群をスチルベノイドからフラボノイドへと変更したと推論できる。