分子進化学が明らかにしたちょっと奇妙な結果は、生物種が変わっても、同一種のタンパク質の進化速度はほとんど同じ、つまり、表現型レベルで急速に変化している生物群でも、何億年間もほとんど変わらない種のものであっても、「同じタンパク質の分子レベルでの進化速度はほとんど同じである」というものである。この知見に従うと、1植物のもつ2種のカルコンシンターゼの進化速度がそれぞれ違うとなどいう安易な解釈はできない。当然の話であろう。では何が起こったか?
先に何度か述べたが、植物においては、遺伝子の重複だけではなくゲノム全体の重複も珍しいことではない。こうして生成した同一機能を持つ遺伝子群のなかでは、少数の遺伝子が機能を失ったとしても生命の維持に支障がないため、いわゆる偽遺伝子と呼ばれる機能を失った遺伝子として保持され続けることが知られている。こうした偽遺伝子では、生物にとって不利な進化が起こっても淘汰が働かないため進化速度が非常に高くなる。通常、偽遺伝子とは、遺伝子として働いていたものが機能を失った場合を指すことが多いが、転写レベルでの発現が確認されている例においては、翻訳産物が機能を維持しているか維持していないかにかかわらず、タンパク質として発現している可能性は存在する。この場合偽遺伝子として捉えるより、むしろアイソザイムグループとして見た方が良いのかもしれない。
では、異常なカルコンシンターゼの進化速度は、タンパク質が持ちうる進化速度の範囲内に収まるのだろうか。異常さが際立っているシロイヌナズナのカルコンシンターゼと、モクマオウの正常なカルコンシンターゼを選んで解析を行うと、
naa=375、 daa=218
という値が得られる。つまり375アミノ酸座位に対して218カ所で変異が起こっているというわけである。この値から進化速度を計算すると kaa = 2.6×10-9という値が得られるのだが、この値は正常なカルコンシンターゼの0.54×10-9より5倍ほど速い。他のタンパク質と比べてみると、リボヌクレアーゼのkaa = 2.1×10-9よりいくぶん早く、もっとも進化の早いフィブリノペプチドのkaa = 8.3×10-9に比べるとかなり遅い。この値から言えるのは、分岐した時間の4分の1程度の間偽遺伝子化し、フィブリノペプチド並みの速度で進化をしたとすれば、シロイヌナズナカルコンシンターゼの異常な進化速度は説明可能となるし、これらの配列を除外したことの合理性が一応は説明できることになる。
次にブドウについて見てみよう。西洋ブドウにおいては、CHS/STSに属する72本のアミノ酸配列がUniPlotでの検索で得られる。これらについてUPGMA法で樹形図を描くと下に示す図が得られる。

これは一応示しただけの図であり、小さすぎて何も分からない。ただ、全体としてこんな形になるよというだけである。だが、気になる特徴がないわけでもない。ブドウは高等植物でありながらSTSをもつ少数派に属するのだが、この植物に存在するCHS/STSグループの中72本のでCHSと比定されるアミノ酸配列は上から8本のみであり、残りの62本はSTSと比定されている。何故ブドウにはこんなにたくさんのSTS の仲間が存在するのか。分からない。しかし、ブドウにSTSが存在するが故にレスベラトロールが生合成され、ワインはとても健康に良いというお伽話が成立している訳である。ここで、アラビドプシスの場合と同様に、これら72本の集合の中からタンパク質として発現している配列を含めて、CHS群から3本、STS群から3本、そして遠縁に当たるらしいF6I151、F6CV80の計8本を選び、UPGMA法で樹形図を描くと下の図が得られるのだが、この図は上の図の簡略版であり当たり前でどうということはない。
この8本にアラビドプシスの場合と同じく、タンパク質レベルでの発現が知られている4本のCHS(アルファルファ、アサ、オトギリソウ、クズ)と5本のSTS(ブドウ、ピーナッツ、ストローブマツ、ヨーロッパアカマツ、およびミカズキゼニゴケ)を加えて解析し、UPGMA法で描くと樹形図は次のようになる。
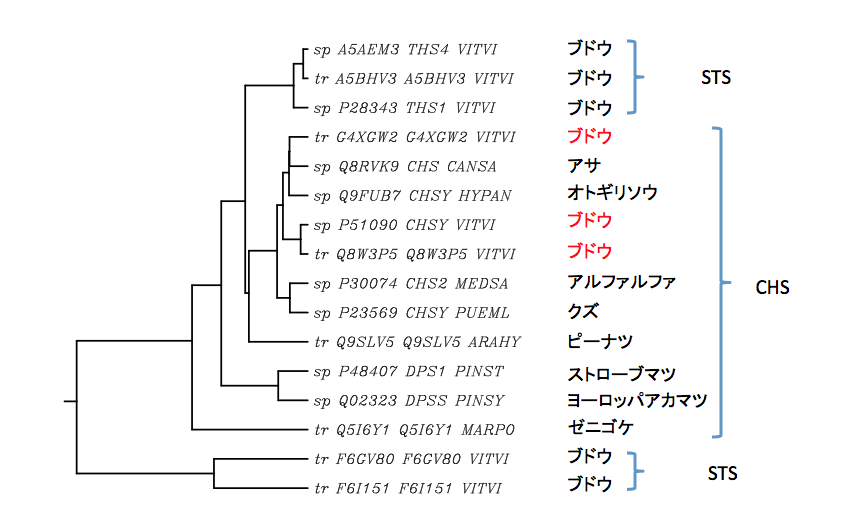
この系統樹はかなり面白い。常識的にこの図を解読するとすれば、高等植物のSTSも、ミカヅキゼニゴケのCHSであるQ5I6Y1から4.7億年ほど前に分岐した後、ゲノム重複によって出現した複数のCHSの一部が、復帰突然変異を起こしながら形成されていったと読み解いてよいだろう。ただ、遠縁に当たるらしいF6I151、F6CV80 については、余りにも分岐した時代が遠過ぎて、現在の知識では説明がつかない。ここまでの議論、CHSとSTSを時には同じに扱い、時には分けて扱うという批判はあり得ると思うが、彼らがそういう関係にあるということもまた事実であろう。
